Ensemble查找启动子

网址:https://www.360docs.net/doc/307809818.html,/index.html
1.
2.
3.
4.
11.查找启动子
2012年3月18日星期日
1:55
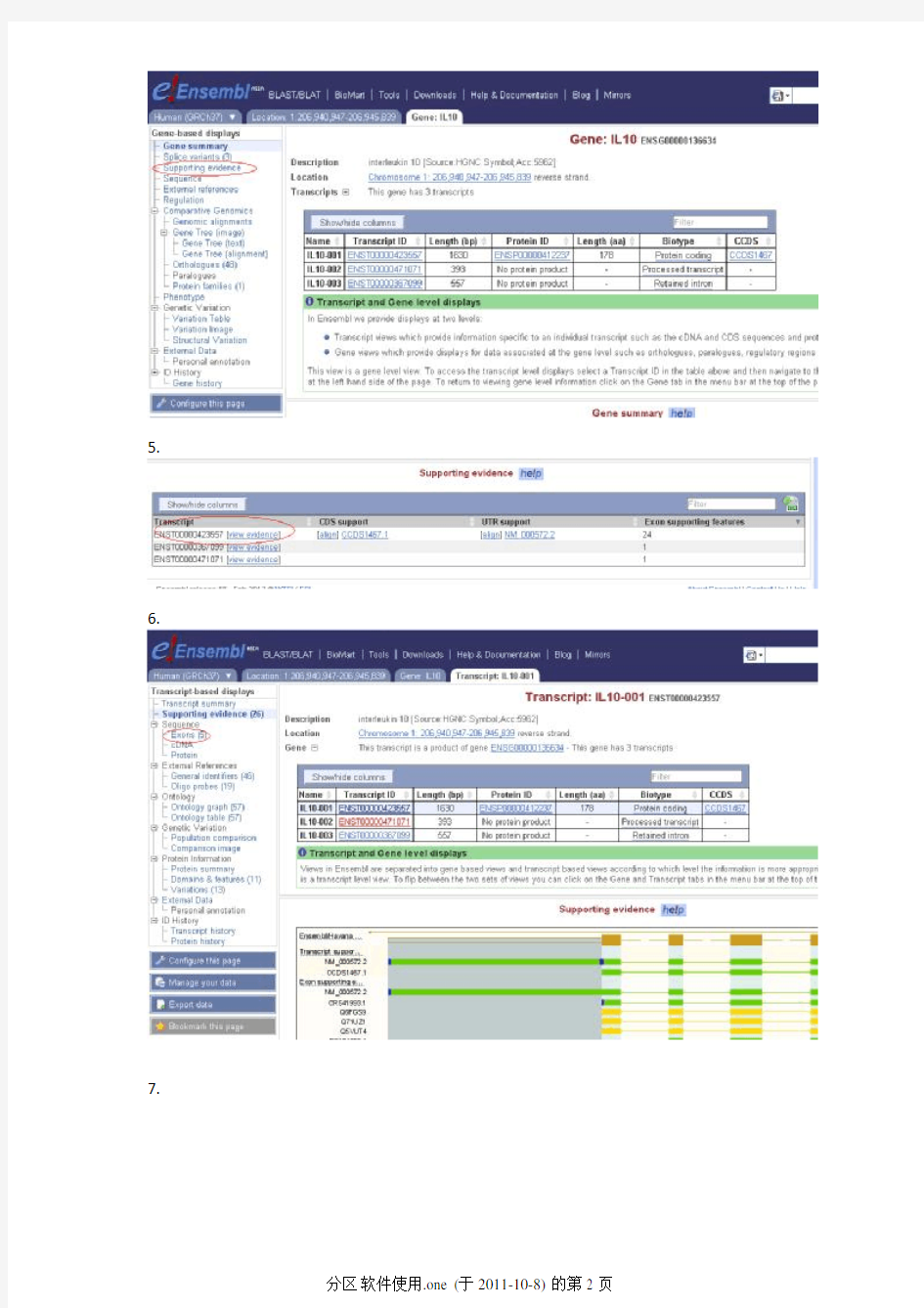
5.
6.
7.
7.
8.
9.
系统集成项目管理规范
省级中心系统集成项目管理规范 为规范本公司系统集成的管理,形成一套行之有效的规范化的工作方法,提高工作效率,明确员工的工作职责,保证工程的质量,特制定本规范。 一、工程组织结构 省级中心工程的系统集成工作采用项目负责制,即由部门负责人指定、报公司批准确定一名项目经理。 项目经理接受任命后,必须与用户项目组协调,确定用户现场项目负责人,制订出可行的工作进度表。根据工程的情况,划分用户项目负责人的任务,由项目经理提名,部门负责人批准配备工程参与人员组成一任务小组,任务小组可以由一名或多名人员组成。项目经理也可以作为工程参与人员。 1.1、项目经理的主要职责: 1)与用户和公司进行工程总协调。 2)计划工程进度,划分用户项目负责人的任务。 3)负责工程参与人员的配备并安排的实施过程,负责工程的进度、包括住宿等。 4)对用户所提出要求的响应。 5)制作和管理工程文档。 6)协调解决工程实施过程中出现的不可预测的问题。 7)向部门和公司领导汇报工作进度。 8)负责监督和考核工程参与人员的工作。
9)保证项目按合同期限和技术要求完成,承担完成工程目标的责任。 1.2、任务小组负责人的主要职责: 1)依照本规定,完成项目经理安排的任务。 2)向项目经理汇报工作进展情况,反馈用户的要求与意见。 3)负责任务准备期和实施期与用户的协调工作。 4)安排本任务小组成员的分配实施工作。 5)制作更新与任务相关的工程文档。 6)解决工程实施过程中出现的不可预测、妨碍进度的因素。 7)承担完成所接受任务的责任。 二、工程工作流程 在立项阶段,根据公司签订的合同,明确项目背景和技术方案,由部门负责人任命项目经理,下达《项目计划书》。 在计划阶段,由项目经理制订出的工程项目计划,划分工程任务,人员配备要求,确认用户项目负责人,并报部门负责人批准。 在准备阶段的主要工作包括设备采购、确定中心机房设备平面位置、中心机房拓扑图等方案准备。 在实施阶段的主要工作包括运输设备、设备到货验收、主机安装调试、数据库安装调试、网络设备安装调试以及其他设备安装调试。
一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST 序列比对等
一步一步教你使用NCBI 查找DNA、 mRNA、cDNA、Protein、promoter、引 物设计、BLAST 序列比对等 最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用 BLAST 进行序列比对……,这些问题在NCBI 上都可以方便的找到答案。现在我就结合我自 己使用NCBI的一些经历(经验)跟大家交流一下BCBI 的使用。希望大家都能发表自己的使 用心得,让我们共同进步! 我分以下几个部分说一下NCBI 的使用: Part one 如何查找基因序列、mRNA、Promoter Part two 如何查找连续的mRNA、cDNA、蛋白序列 Part three 运用STS 查找已经公布的引物序列 Part four 如何运用BLAST 进行序列比对、检验引物特异性 特别感谢本版版主,将这个帖子置顶! 从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我 投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友! 请大家对以下我发表的内容提出自己的意见。关于NCBI 其他方面的使用也请水平较高 的战友给予补充 First of all,还是让我们从查找基因序列开始。 第一部分利用Map viewer 查找基因序列、mRNA 序列、 启动子(Promoter) 下面以人的IL6(白细胞介素6)为例讲述一下具体的操作步骤 1.打开Map viewer 页面,网址为:https://www.360docs.net/doc/307809818.html,/mapview/index.html 在search 的下拉菜单里选择物种,for 后面填写你的目的基因。操作完毕如图所示:
基因启动子分析基本流程
“螺旋讲堂”2008 年第十一课----“基因启动子分析基本流程”
“螺旋讲堂”2008年第十一课----“基因启动子分析基本流程”
螺旋 亲爱的螺友们,大家好!欢迎光临螺旋讲堂,很高兴有机会和大家相聚螺旋网,让 我们一同在讨论中学习,在交流中成长! 分子生物学发展迅猛,新方法新技术新发现层出不穷,但是我想,我们的基础研究从 某种意义上来说,可以简单的分为两大部分,一个是基因的表达,另一个是基因的功能。当 然,这个基因的概念现在已经不仅仅是指编码蛋白的 DNA 序列了。 我们这期主要探讨基因的表达。而转录调控在基因表达中占有很重要的地位。基因 的转录调控机制非常复杂,这些理论有机会我们再详细探讨,这里就不多介绍了,我们主要 谈一下对于一个新的基因,如何开始他的转录调控研究,第一步到底该怎么做呢? 这里提供一些简单的入门级别的方法,希望对大家有用。相信还有更多更好更实用 的方法,也希望螺友们能够拿出来和大家分享,共同进步! 本次讲座共分为五个部分主要是讲第一部分,因为这个一般的文献和书籍都很少有 详细说明.
一:克隆目的基因基本启动子序列 我们都知道, 基因的基本启动子一般是在基因转录起始位点上游, 当一个基因在没有 确定其转录起始位点的时候,我们假定 NCBI 上提交的序列就是他的完整转录本,那么他的 第一个碱基就是他的转录起始位点。而基因的基本启动子一般就是在转录起始位点的上游 2000bp 左右和下游200bp 左右,当然,这个是一般情况,具体问题还要具体分析.尤其现在发 现一般的基因都是有几个转录起始位点的. 我们通过该基因 mRNA 序列和基因组序列 BLAST, 就能够在染色体上找到这段基因 组序列。我这里用 human 的 AGGF1基因做个例子给大家具体演示一下.
https://www.360docs.net/doc/307809818.html,
系统集成项目管理
系统集成项目管理 第一章信息系统概述 1.1 信息与信息系统 1.1.2 信息系统 信息系统概念:信息系统是与信息加工、传递、存储、利用有关的系统 信息系统一般包括:(1)数据处理系统(2)管理信息系统(3)决策支持系统(4)办公自动化系统 1.数据处理系统:主要功能是将输入的数据信息进行加工、整理、计算各种分析指标,变为易于被人们接受的信息,并将处理后的信息进行有序的存储,随时通过外部设备输给信息使用者。它包括:对数据进行收集、存储、传输、变换的过程。 2.管理信息系统:是为了适应现代化管理的需要,它研究系统息处理和决策的整个过程,它由人、计算机、通信设备等硬件和软件构成,能进行管理信息的收集、加工、存储、传输和维护使用。 3.决策支持系统:包括结构化、半结构化(无经验可询)和非机构化(人机对话) 4.办公自动化系统 1.2 信息系统工程 1.信息系统工程的几个阶段: 按照生命期来讲,信息系统工程包括:立项、规划、建设、应用、维护几个阶段。 2.信息系统工程的容: (1)信息网络系统
(2)信息资源系统 (3)信息应用系统(必会) 信息应用系统的生命期包括 4 个阶段:产生、开发、运维、消亡(必会)1)产生阶段也成为信息系统的概念阶段,需求分析阶段 2)开发阶段:开发阶段分为以下几个子阶段: a) 总体规划 b) 系统分析 c) 系统设计 d)系统实施 e)系统验收 3)运维阶段:信息系统验收通过,正式交给客户后,系统进入运行阶段。运维阶段维护的四种类型(就是鱼丸) 纠错性维护(检修) 适应性维护(升级) 完善性维护(提升功能,工作量最大) 预防性维护(工作量最小) 4)消亡阶段 (4)信息系统的开发法: 1)结构化法(需求明确,是最成熟,最广泛的开发法之一) 2)快速原型法(适用于需求模糊,结构性较差的项目)包括进化型原型和抛弃型原型3)企业系统规划的法:其目标是提供一个信息系统规划,用以支持企业短期长期的要求
DNA启动子概述
启动子概述 启动子是DNA链上一段能与RNA聚合酶结合并能起始mRNA合成的序列,它是基因表达不可缺少的重要调控序列。启动子是一段位于结构基因5’-端上游区的DNA序列,能活化RNA聚合酶,使之与模板DNA准确地结合,并具有转录起始的特异性。基因的特异性转录取决于酶与启动子能否有效地形成二元复合物。启动子分三类:启动子Ⅰ、启动子Ⅱ、启动子Ⅲ.只有启动子Ⅱ指导mRNA的转录。真核生物启动子Ⅱ由两大部分组成:上游元件(upstream element)和启动子核心(core promoter)。上游元件与转录的效率有关;启动子核心包括3部分:TATA 盒、起始子(initinator)及下游元件(downstream element)。TATA盒为转录调控因子包括各种调节蛋白的结合区,与转录起始位点的精确选择及转录有关,起始子是转录起始所必须,下游元件作用尚不清楚。原核生物启动子区范围较小,包括TATAAT区(Pribnow区)及其上游的TTGACA区。 启动子是一段提供RNA聚合酶识别和结合位点的DNA序列,位于基因上游。启动子具有如下特征: 1序列特异性。在启动子的DNA序列中,通常含有几个保守的序列框,序列框中碱基的变化会导致转录启动活性的改变。 2方向性。启动子是一种有方向性的顺式调控元件,有单向启动子和双向启动子两类。 3位置特性。启动子一般位于所启动转录基因的上游或基因内的前端。处于基因的下4种属特异性。原核生物的不同种、属,真核生物的不同组织都具有不同类型的启动 没有启动子,基因就不能转录。原核生物启动子是由两段彼此分开且又高度保守的核苷酸序列组成,对mRNA的合成极为重要。启动子区域:(1)Pribnow盒,位于转录起始位点上游5—10bp,一般由6~8个碱基组成,富含A和T, 故又称为TATA盒或—10区。启动子来源不同,Pribnow盒的碱基顺序稍有变化。(2)—35区,位于转录起始位点上游35bp处,故称—35区,一般由10个碱基组成。 质粒设计时都需要加入启动子序列,以保证目的基因的表达。启动子可分为诱导型启动子和组成型启动子两大类,后者包括CMV,SV40,T7,pMC1,PGK启动子等。一下介绍几个常见的启动子。 (1)U6启动子 U6是二型启动子,一般发现是启动小片段,不带PolyA尾的序列。由Ⅲ类RNA聚合酶启动子U6启动子转录产生shRNA,经剪切后产生成熟siRNA,产生干扰效果。这一类 启动子在腺病毒和慢病毒干扰载体的构建中应用很多。U6更多的是用在shRNA的启动,来达到敲低一个基因的作用。
找一个基因的启动子
1、UCSC (1)网址:https://www.360docs.net/doc/307809818.html,/cgi-bin/hgNear 在Genome里选择物种,比如human,search里输入你的基因名PTEN,点击Go (2)出现新的页面,看到“Known Gene Names”下面的PTEN了吧,点它 (3)又回到了和(1)类似的页面,此时,点击sequence (4)出现一个新的页面,选中promoter,同时可以输入数值修改具体的序列区域,比如Promoter including 2000 bases upstream and 100 downstream,即表示启动子-2000~+100区域 (5)点击“get sequence”,出现页面中最上面的序列“>uc001kfb.1 (promoter 2000 100) PTEN - phosphatase and tensin homolog”就是你要的人PTEN启动子-2000~+100区域的序列了 2、Ensembl (1)网址:https://www.360docs.net/doc/307809818.html,/index.html 在“Search Ensembl“标题下search后的下拉框中选中物种名homo sapiens(人),for框中输入基因名PTEN,点击Go (2)出现的新页面中比较乱,但不要管它,直接寻找“Ensembl protein coding gene ”字样的,对,也就是第二个,点击它 (3)新出现的页面也很乱,不过依然不用管它,看到左侧有点肉色(实在不知道怎么描述了)的那些选项了吗,对,就是“Your Ensembl”下面那一堆,在里面找“Genomic sequence”,点它 (4)现在的界面就一目了然了,在“5' Flanking sequence”中输入数值确定启动子长度(默认为600),比如1000,点击update; (5)出现的序列中,标为红色的就是基因的外显子,红色之间黑色的序列就是内含子,而第一个红色自然就是第一外显子了,那么从开始的碱基一直到第一个红色的碱基间自然就是启动子-1000~+1的序列啦 这样,你不仅查到了启动子,连它的外显子、内含子序列也全部搞定了
如何查找一个基因的启动子序列
定义:启动子是参与特定基因转录及其调控的DNA序列。包含核心启动子区域和调控区域。核心启动子区域产生基础水平的转录,调控区域能够对不同的环境条件作出应答,对基因的表达水平做出相应的调节。一般查阅外文文献,老外从转录起始位点(Transcription Strart Site,TSS,记为+1位)开始上溯2K -3K的区间算做是启动子 区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于启动子范围。 票数 Do One Thing, And Do It Well. mybbff edited on 2005-07-22 08:41 ? ? ? ? 2005-05-07 11:23 分享 分享到哪里? ? ?
??? ? ? ? ? 下面以BCL-2基因为例,查找查找该基因的启动子区域,首先要找到该基因的基因组序列。去NCBI吧,在Search的下拉菜单里找到Gene,在检索项里输入Bcl-2,检索第一项就是bcl-2 for human,点进去看看啥样。。。 票数 Do One Thing, And Do It Well. ??
???2005-05-07 11:29 分享 分享到哪里? ? ? ? ? ? ? 首先你可以看到该基因的参考序列(reference sequence),然后看到bcl-2的位置和基因组背景。bcl-2上游是 PHLPP,下游是FVT1基因。在这个长长的网页的最后是已经注册的Bcl-2基因的信息。
票数 Do One Thing, And Do It Well. Revelation edited on 2005-05-07 11:59 ? ???2005-05-07 11:35 分享 分享到哪里? ? ? ? ? ? ? 看到基因组序列了么,点进去,根据序列信息自己就能定位转录起始位点,上游就是promoter了,简单吧。不!我觉得麻烦。有更简单的方法么?有!注意到在网页的开头有这么个链接么?HGNC:990
人全外显子组序列捕获及第二代测序
人全外显子组序列捕获及第二代测序 概述 外显子组是指全部外显子区域的集合,该区域包含合成蛋白质所需要的重要信息,涵盖了与个体表型相关的大部分功能性变异。外显子组序列捕获及第二代测序是一种新型的基因组分析技术:外显子序列捕获芯片(或溶液)可在同一张芯片上以高特异性和高覆盖率捕获研究者感兴趣的目标外显子区域,后续利用Solexa/SOLiD/Roche 454测序直接解析数据。 与全基因组重测序相比,外显子组测序只需针对外显子区域的DNA 即可,覆盖度更深、数据准确性更高,更加简便、经济、高效。可用于寻找复杂疾病(如:癌症、糖尿病、肥胖症等)的致病基因和易感基因等的研究。同时,基于大量的公共数据库提供的外显子数据,我们能够结合现有资源更好地解释我们的研究结果。 目前,SBC提供的外显子组序列捕获芯片是NimbleGen Sequence Capture 2.1M Human Exome Array及Agilent SureSelect Target Enrichment System(Human Exome)。 技术路线 以Nimblegen外显子捕获结合Solexa测序为例加以说明:基因组DNA首先被随机打断成500bp左右的片段,随后在DNA片段两端分别连接上接头。经过PCR库检合格后的DNA 片段与NimbleGen 2.1M Human Exome Array芯片进行杂交。去除未与芯片结合的背景DNA 后,将经过富集的外显子区域的DNA片段洗脱下来。这些DNA片段又随机连接成长DNA片段
后,再次被随机打断并在其两端加上测序接头,经过LM-PCR的线性扩增,在经qPCR质量检测合格后即可上机测序。 外显子组测序的实验流程示意图(https://www.360docs.net/doc/307809818.html,) 生物信息学分析流程图 研究内容 1.外显子组捕获与测序 将基因组DNA随机打断成片段,通过与人全外显子捕获芯片杂交富集外显子区域,通过第二代测序技术对捕获的序列进行测序。 2.基本数据分析 数据产出统计:对测序结果进行图像识别(Base calling),去除污染及接头序列;统计结果包括:测定的序列(Reads)长度、Reads数量、数据产量。 3. 高级数据分析 高级数据分析内容包括: (1)Clean reads序列与参考基因组序列比对; (2)目标外显子区域测序深度分析; (3)目标外显子区域一致序列组装;
系统集成方案.
系统集成(BMS) 一、系统概述 智能化集成系统(IBMS)是一个在技术上、品质管理上、施工管理上都有很高要求的项目,我方特别为这个项目的设计拟定了本系统设计规范说明,以便参与本项目的工作人员能对大楼智能楼宇管理系统的功能、设计及要求有所理解,并确定了系统设计的标准。我方设计根据某综合楼的性质、用途特点,采用先进、成熟的技术对整个大楼的弱电子系统,包括建筑设备管理系统(BAS)、消防自动报警系统(FAS)、公共安全系统(报警、监控系统、门禁系统、停车场管理系统)智能卡应用系统(门禁系统、停车场管理系统),信息引导及发布系统、设备与工程档案管理系统进行统一集成,形成一个统一的、相互关联的、相互协调联动的、在同一平台上运行的综合管理系统,实现楼宇信息的高度共享。 二、设计原则
建筑自动化管理系统的总体设计原则是: 以计算机网络为基础、软件为核心,根据智能化系统工程工作原理,通过信息交换和共享,结合智能建筑的工程建设的一些实际时间经验,将各个具有完整功能的独立分系统组合成一个有机的整体,建立统一的网络管理平台,提高系统维护和管理的自动化水平、协调运行能力及详细的管理功能,能够对各个智能化子系统进行综合管理,满足整个智能化系统预期的使用功能和管理要求,彻底实现功能集成、网络集成和软件界面集成,最大限度地获取系统的综合效益。 三、系统设计方案 1、KITOZER30000建筑自动化管理系统综述 本工程采用广州莱安KITOZER30000建筑自动化管理系统,该在总结多年建筑自动化行业建设经验的基础上,综合研究国外知名的楼宇自动化系统和开发平台并应用最先进的软硬件技术研制成功的。KITOZER30000面向建筑自动化行业、采用子系统集成模式的,集数据采集、网络通信、自动控制和信息管理于一体,是一种可二次开发的监控管理平台软件。 KITOZER30000具有使用简单、性能可靠、速度快、系统开放等特点,高可靠性和高弹性,可广泛应用于智能大厦、智能小区等智能建筑物。 KITOZER30000的总体目标是:“以计算机网络为基础、软件为核心,通过信息交换和共享,将各个具有完整功能的独立子系统整合成一个有机体,实现系统的信息共享,降低系统的运行费用,提高
如何查找一个基因的启动子序列
如何查找一个基因的启动子序列 发表者:刘小丰 (访问人次:6102) 刘小丰收集整理 定义:启动子是参与特定基因转录及其调控的DNA序列。包含核心启动子区域和调控区域。核心启动子区域产生基础水平的转录,调控区域能够对不同的环境条件作出应答,对基因的表达水平做出相应的调节。 区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于启动子范围。 这项搜寻要从UCSC基因组浏览器开始,网址为 https://www.360docs.net/doc/307809818.html,/cgi-bin/hgGateway。以编码pendrin (PDS)的基因为例来说明上述问题。PDS与耳蜗的异常发育、感觉神经性听力下降以及弥散性甲状腺增大(甲状腺肿)有关。 进入UCSC的主页后,在Organism的下拉菜单中选择Human,然后点击Browser。使用者现在到了人类基因组浏览器入口。本例的搜寻很简单:在assembly的下拉菜单中选择Dec. 2001,在position框中键入pendrin,然后点击Submit。返回的页面结果显示一个已知的基因和两个mRNA序列。继续点击mRNA序列的登录号AF030880,出现包含这个mRNA区域的图解概要。为了获得这个区域更清晰的图像,点击紧靠zoom out的1.5X按钮。最后点击页面中部的reset all按钮,使各个路径的设置恢复默认状态。 然而,对于本例的搜寻目的来说,默认设置不是理想的设置。按照视图利用页面底部的Track Controls按纽,将一些路径设置为hide模式(即不显示),其他设置为dense模式(所有资料密集在一条直线上);另一些路径设置为full模式(每个特征有一个分开的线条,最多达300)。在考虑这些路径内究竟存在那些资料之前,对这些路径的内容和表现做一个简要的讨论是必要的,许多这些讨论是由外界提供给UCSC的。下面是对基因预测方法的更进一步讨论,这些信息也可以在其他地方找到。 对于Known Genes(已知基因)和预测的基因路径来说,一般的惯例是以一个高的垂直线或块状表示每个编码外显子,以短的垂直线或块状表示5′端和3′端非翻译区。 起连接作用的内含子以非常细的线条表示。翻译的方向由沿着细线的箭头指示。 Known Genes来自LocusLink内的mRNA参照序列,已经利用BLAT程序将这些序列与基因组序列进行比对排列。 Acembly Gene Predictions With Alt-splicing路径是利用Acembly程序将人类mRNA 和EST序列数据与人类基因组序列进行比对排列而来的。Acembly程序试图找到mRNA与基因组序列的最好的比对排列以及判断选择性剪接模型。假如有多于1个的基因模型具有统计学意义,则它们都全部显示出来。有关Acembly的更多信息可以在NCBI的网站找到(https://www.360docs.net/doc/307809818.html,/IEB/Research/Acembly/)。 Ensembl Gene Predictions路径由Ensembl提供。Ensembl基因通过许多方法来预测,包括与已知mRNA和蛋白质进行同源性比较,ab initio基因预测使用GENSCAN和基因预测HMMs。 https://www.360docs.net/doc/307809818.html,/ensembl/ Fgenesh++ Gene Predictions路径通过寻找基因的结构特征来预测基因内部的外显子,例如剪接位点的给位和受位的结构特征,利用一
基因启动子分析
基因启动子分析 一:克隆目的基因基本启动子序列 我们都知道,基因的基本启动子一般是在基因转录起始位点上游,当一个基因在没有确定其转录起始位点的时候,我们假定NCBI上提交的序列就是他的完整转录本,那么他的第一个碱基就是他的转录起始位点。而基因的基本启动子一般就是在转录起始位点的上游2000bp左右和下游200bp左右,当然,这个是一般情况,具体问题还要具体分析.尤其现在发现一般的基因都是有几个转录起始位点的. 我们通过该基因mRNA序列和基因组序列BLAST,就能够在染色体上找到这段基因组序列。我这里用human的AGGF1基因做个例子给大家具体演示一下. 1 首先需要在NCBI里面查找到AGGF1基因的mRNA序列,这个我想大家都应该很清楚,如下图.
2 然后就是用这段mRNA序列和人类的基因组序列BLAST 3 BLAST得到了很多结果,我们往往选择最上面那个最匹配的结果。
4 点击之后就可以看到下图,这个基因的14个外显子和13个内含子在5号染色体上的位置一目了然,第一个外显子在上面,说明这个基因在染色体上是正向的,基本启动子就应该在第一外显子上面,我用红色的方框标明了。 5 大家有没有注意到左上方有个数据框,我把数值改为76,360K 到 76,362.200 ,刚好2200BP,包括了第一个外显子的前200BP左右. 然后点击红色框标明的Download/view sequence.
6 然后就到了这个界面, Sequence Format 选择GenBank, 然后点击 Display. 就得到我们所需要的序列了. 7 这里我们可以看到1989到2201是AGGF1的mRNA序列,说明我们的确找到了该基因5'非翻译区的上游启动子序列.建议将这2200bp都克隆下来. 以上的步骤就是基因基本启动子的查找,其实还有很多调控序列是在基因内含子区域或者是基因的3'非翻译区等,序列查找的步骤和上面是一样的.
系统集成项目管理系统使用说明
系统集成项目管理系统使用说明 1.系统简介 为规范系统公司的项目管理,实现公司各部门对工作量和进度的反馈,特开发《系统集成项目管理》系统。该系统依托于公司的Notes平台,用于系统公司各项目的监控。 系统将项目分为立项、提案、投标、开发、工程、结项、维护这几个阶段,项目可以从立项、提案、投标、开发、工程中的任一阶段开始。项目开始后,系统会自动为其建立主项目单,在这个主单上关联项目的各个阶段。一个项目可以包括经历多次提案、投标、开发、工程过程申请,结项后可以有多次维护。 项目的售前负责人、项目负责人、工程负责人可以给项目成员建立任务单。 项目中需要提供业主信息、合作伙伴信息,如需施工还需提供工程商信息。这些信息以分公司为单位归类,新信息可以使用特定的按钮创建,也可以在项目开始时在申请信息中直接填写来创建。 2.立项
2.1输入和输出 2.1.1输入 序号名称描述模板 1 业主信息无 2 合作伙伴信息无 3 立项阶段需求描述项目立项阶段的基本需求、要点 难点、要求输出成果物和要求完成日 期等 无 2.1.2输出 序号名称描述模板 1 初步解决方案无 2 概算清单无 3 PPT演讲稿无 2.2角色与职责 角色职责 直接主管1、审核概算清单 售前负责人1、分派立项阶段的任务 2、提交初步解决方案、概算清单、PPT演讲稿等输出成果物 3、移交审核过的初步解决方案、概算清单、PPT演讲稿等输出成果物 受理人1、审核立项申请 2、指定售前负责人 申请人1、提出需求,提交立项申请 2、确认立项结束 2.3流程图
2.4流程描述 提出需求,提交立项申请 责任角色:申请人 1.申请人根据所属分公司,选择相应的业主信息、合作伙伴信息。 2.申请人整理立项阶段需求,提交立项申请。 补充说明: 1.业主信息、合作伙伴信息与各分公司关联,先选择所属分公司,才能显 示与之对应的业主、合作伙伴。 2.对于没有入库的业主、合作伙伴信息,需要先创建。创建方式有两种: 一种是使用表单上方的“创建业主信息”“创建伙伴信息”按钮,如下图; 一种是选择了所属分公司后,直接在申请表单填写新的业主、合作伙伴 信息,提交后新信息将自动入库。
软件系统集成与整合的常见方式
企业在信息化的过程中会根据自身的需求构建各种软件系统,如:网站、OA、CRM、订单系统、采购系统、库存管理、财务系统等,由于所需的软件系统一般是逐步构建和投入使用的,构建的时间、所采用的技术等都不一样,软件系统的也很难做到完全由一家供应商提供。如果企业的多个系统之间存在信息传递和数据交换,软件系统之间的集成和整合就势在必行,如:OA中需要访问CRM的数据、CRM需要访问订单系统的数据;CRM和订单系统都存在客户信息的维护管理,为了保证数据的唯一和准确、同时减少维护的工作量,最好是只在一个系统中进行管理和维护等等,那么软件系统集成和整合的方式常见的有哪些呢? 一、软件系统功能完全融合在一个系统中 A、方式描述 将多个系统融合在一个系统中,统一账号和权限的管理,统一应用的管理,最终以一个独立的软件系统存在。如果这种方式所需的时间和成本比较低,该模式在管理和使用上对最终用户更加方便。 B、应用场合 1、以某一个软件系统为主、需要整合的功能比较简单; 2、软件系统是以定制开发为主的,后续需要定制开发新的功能; 3、一般由同一个软件供应商提供服务; C、优势 所有功能都在一个系统中,节省资源,方便管理和维护,系统之间的信息传递及时快捷,功能完整性比较好。 D、不足 软件服务商需要有较强的开发能力,周期比较长,需要对所有系统都非常熟悉,对已有系统的扩展性要求比较高(否则代价高、造成已有系统的不稳定)。 二、软件系统间以接口方式相互调用 A、方式描述 企业存在多个各自独立的软件系统,系统之间调用彼此的接口进行数据的交换和信息的传递。如,OA系统中读取订单系统的销售数据进行业绩统计和绩效管理,OA系统中费用报销流程的数据需写入财务系统,网站中客户下单的信息需写入到OA系统进入订单审批流程,网上支付银行接口的调用等。 一般在技术上会以API接口、web service接口、直接访问数据库接口等方式实现,优秀的软件系统一般都有设计良好的外部接口,直接访问数据库不是最好的解决方案。 B、应用场合 1、多个软件系统独立存在,每个系统的都占有比较重要的地位,软件系统可能由不同的供应商提供。 2、系统之间需进行数据的交换和信息的传递,企业的某些业务需要经过多个系统的处理才能完整的完成。
如何查找一个基因的启动子序列
如何查找一个基因的启动子序列 关键词:基因启动子序列软 件 定义:启动子是参与特定基因转录及其调控的DNA序列。包含核心启动子区域和调控区域。核心启动子区域产生基础水平的转录,调控区域能够对不同的环境条件作出应答,对基因的表达水平做出相应的调节。 区域:启动子的范围非常大,可以包含转录起始位点上游2000bp,有些特定基因的转录区内部也存在着转录因子的结合位点,因此也属于启动子范围。 这项搜寻要从UCSC基因组浏览器开始,网址为https://www.360docs.net/doc/307809818.html,/。以编码pendrin (PDS)的基因为例来说明上述问题。PDS与耳蜗的异常发育、感觉神经性听力下降以及弥散性甲状腺增大(甲状腺肿)有关。 进入UCSC的主页后,在Organism的下拉菜单中选择Human,然后点击Browser。使用者现在到了人类基因组浏览器入口。本例的搜寻很简单:在assembly的下拉菜单中选择Dec. 2001,在position框中键入pendrin,然后点击Submit。返回的页面结果显示一个已知的基因和两个mRNA序列。继续点击mRNA序列的登录号AF030880,出现包含这个mRNA区域的图解概要。为了获得这个区域更清晰的图像,点击紧靠zoom out的1.5X按钮。最后点击页面中部的reset all按钮,使各个路径的设置恢复默认状态。 然而,对于本例的搜寻目的来说,默认设置不是理想的设置。按照视图利用页面底部的Track Controls按纽,将一些路径设置为hide模式(即不显示),其他设置为dense模式(所有资料密集在一条直线上);另一些路径设置为full 模式(每个特征有一个分开的线条,最多达300)。在考虑这些路径内究竟存在那些资料之前,对这些路径的内容和表现做一个简要的讨论是必要的,许多这些讨论是由外界提供给UCSC的。下面是对基因预测方法的更进一步讨论,这些信息也可以在其他地方找到。 对于Known Genes(已知基因)和预测的基因路径来说,一般的惯例是以一个高的垂直线或块状表示每个编码外显子,以短的垂直线或块状表示5′端和3′端非翻译区。 起连接作用的内含子以非常细的线条表示。翻译的方向由沿着细线的箭头指示。 Known Genes来自LocusLink内的mRNA参照序列,已经利用BLAT程序将这些序列与基因组序列进行比对排列。
外显子捕获结题报告
外显子捕获结题报告2010-11-22
内容 1 项目信息 (1) 2 工作流程介绍 (2) 2.1 Agilent液相捕获平台 (2) 2.2 NimbleGen 液相捕获平台 (3) 2.3 生物信息分析流程 (4) 3 分析报告 (5) 结果 (5) 3.1 标准生物信息分析 (5) 3.1.1 数据产出统计 (5) 3.1.2 目标区域单碱基深度分布图 (6) 3.1.3外显子捕获测序的均一性 (7) 3.1.4一致序列组装和SNP检测 (7) 3.1.5 SNP注释 (8) 3.1.6插入/缺失(indels)检测 (9) 3.1.7插入/缺失(indels)注释 (9) 3.2个性化分析 (9) 3.2.1氨基酸替换预测 (9) 3.2.2群体SNP检测和等位基因频率估计 (12) 3.2.3孟德尔遗传病分析 (13) 3.2.4 NGS-GW AS 分析 (14) 3.2.5正向选择信号的检测 (14) 4 数据分析方法说明 (15) 4.1信息分析软件及常用参数介绍 (15) 4.2参考数据库 (16) 4.3数据文件格式 (17)
1 项目信息 PROJECT NAME CONTRACT NUMBER SAMPLE INFORMATION Species Information Genome Information Additional Information CUSTOMER INFORMATION PI Contact Person Company Name Contact Methods Name Tel E-mail Name Tel E-mail CONTACT INFORMATION (BGI) Sales Information Name Tel E-mail Name Tel E-mail Customer Service Name Tel E-mail Name Tel E-mail PROJECT DIRECTOR APPROVAL THE RESULTS HAVE BEEN APPROVED AND CAN BE SUBMITTED Signature: Date:
服务器系统集成管理平台用户手册
一、系统简介 服务器系统集成管理平台主要的功能就是对服务器的数据信息进行基本的管理,管理之后保证对数据的信息进行基本的集成的操作,对各个服务器的数据信息进行基本的处理和操作的管理,分配基本的数据信息和端口信息。 二、系统功能 1. 系统管理员登录 在地址栏输入地址,进入系统登录页面。登录界面共包括两部分内容:登录信息和控制按钮。如下图所示: 输入信息:用户名称、登录密码
控制按钮:登录、退出 2.系统基本配置 2.1系统基本配置 系统基本配置包括对设备的网卡及相关参数的设置。界面如下图所示: 管理员可以根据实际网络情况,合理配置网络参数。管理员对网络参数的设置,无需设备重启或服务重启,将会立即生效。例如:现在将系统的主机名设为:;网关:192.168.1.1;:202.101.172.46 202.101.172.47。具体如下图所示:
系统的网卡属性拥有普通和两种模式。普通模式即网卡的正常模式;模式是指以两块网卡提供冗余功能,工作方式是主备的工作方式,也就是说默认情况下只有一块网卡工作,另一块做备份。界面如下图所示: 2.2基本输出设置 二次日志记录加强了原始日志材料的防伪防杜撰功能,通过对比保存于两台日志服务器上的日志材料,可以准确可靠的进行故障鉴定和责任认定,需要对日志服务器参数设置,包括日志服务器地址设置,和“启动/停止”设置,见下图: 2.3时间同步设置 系统的策略控制提供日期/时间控制的策略因子。所以日期/时间的准确性相当重要,而帕拉迪服务器安全管理系统提供的时间同步功能能够与世界标准时间相一致,保证了策略控制的准确性。但使用时间同步功能的同时必须保证系统能和网络相连通。界面如下图所示:
06-重测序、外显子组试卷答案.pdf
一、名词解释 1.比对将测序序列比对到参考基因组序列 2.单核苷酸多态性主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多 态性 3.三体家系样本,父亲,母亲,和孩子家系外显子组中的一组家系样本 4.小的核苷酸的插入缺失 5.拷贝数变异是由基因组发生重排而导致的,一般指长度为1 kb以上的基因组大片段 的拷贝数增加或者减少,主要表现为亚显微水平的缺失和重复 二、填空题 1.bwa GATK 2.1% 3. 全基因组重测序全外显子组区域捕获测序 4. snp indel CNV SV 5. 5 50X 6. 1.5 3.5 7.0.1% 1% 8.液氮冻存-80冰箱冻存 9.血液 10.Agilent SureSelect All Exon V4 (+ UTR), NimbleGen SeqCap EZ Human Exome Library v3.0, Illumina TruSeq Exome Enrichment Kit 51M(71M), 64M, 62M 三、问答题 1. 建库:将基因组DNA经Covaris破碎仪随机打断成长度为180-280bp的片段,末端修复和加A 尾后在片段两端分别连接上接头制备 DNA 文库。 捕获:带有特异index的文库pooling后与多达543,872个生物素标记的探针进行液相杂交,再使用带链霉素的磁珠将20,965个基因的334,378个外显子捕获下来。 扩增及测序:经PCR线性扩增后进行文库质检,合格即可进行测序。 2. 人类85%的疾病位点位于编码区 域外显子组可以提供更深的测序 深度外显子组花费更低 3. 数据质控,比对到参考基因组,去重复重校正,预测个体snp和indel,预测体细胞突变,预测CNV和SV,候选位点注释 4. 常染色体显性遗传 常染色体隐形遗传 伴X 染色体显性遗传 伴X 染色体隐性遗传 伴Y 染色体遗传 找Denovo mutation,只在患病孩子有,不在健康父母里存在的位点 5.
系统集成项目管理规范(网络下载)
系统集成项目管理规范 目录 一、总则 (2) 第一章工程组织结构 (3) 第二章工程工作流程 (4) 第三章工程管理与考核 (4) 第四章工程文档管理 (5) 第五章工程准备管理 (6) 第六章工程实施管理 (7) 第七章工程验收管理 (8) 第八章工程维护管理 (9) 第九章出差管理 (10) 第十章培训体系 (10)
一、总则 第1条为规范本公司系统集成的管理,形成一套行之有效的规范化的工作方法,提高工作效率,明确员工的工作职责,保证工程的 质量,特制定本规范。 第2条鉴于本公司的实际工作分工,本规范所指的系统集成并不包括项目的前期方案设计和应用软件的开发和维护。
第3条本规范的主要内容包括:工程组织结构、工程工作流程、工程管理与考核、工程准备管理、工程准备管理、工程实施管理、 工程验收管理、工程维护管理、出差管理、培训体系共十大部 分。 第一章工程组织结构 第1条工程的系统集成工作采用项目负责制,即由部门负责人指定、报公司批准确定一名项目经理。 第2条项目经理接受任命后,必须与用户项目组协调,确定用户现场负责人,制订出可行的工作进度表。根据工程的情况,划分子系统,并针对不同的子任务,由项目经理 提名,部门负责人批准配备工程参与人员组成一任务小组,任务小组可以由一名或 多名人员组成。项目经理也可以作为工程参与人员。 第3条针对不同的子任务,由项目经理指定一个工程参与人员为任务小组负责人。 第4条项目经理的主要职责: 1)与用户和公司进行工程总协调。 2)计划工程进度,划分工程的子任务。 3)负责工程参与人员的配备并安排的实施过程,负责工程的成本、包括住宿、长途交通费、市内交通费等。 4)对用户所提出要求的响应。 5)制作和管理工程文档。 6)协调解决工程实施过程中出现的不可预测的问题。 7)向部门和公司领导汇报工作进度。 8)负责监督和考核工程参与人员的工作。 9)保证项目按合同期限和技术要求完成,承担完成工程目标的责任。 10)承担用户满意度责任。 第5条任务小组负责人的主要职责: 1)依照本规定,完成项目经理安排的任务。 2)向项目经理汇报工作进展情况,反馈用户的要求与意见。 3)负责任务准备期和实施期与用户的协调工作。 4)安排本任务小组成员的分配实施工作。 5)制作更新与任务相关的工程文档。 6)解决工程实施过程中出现的不可预测、妨碍进度的因素。 7)承担完成所接受任务的责任。 8)承担用户满意度的责任。 第6条工程参与人员的主要职责: 1)依照本,与任务小组负责人协同完成工程安排的任务。 2)及时反馈用户的要求与意见。 3)在任务实施期与用户的协调工作。
miRNA基因和编码基因启动子区核小体定位分析
2010年 第55卷 第14期:1335 ~ 1340 https://www.360docs.net/doc/307809818.html, https://www.360docs.net/doc/307809818.html, 英文版见: Liu H D, Zhang D J, Xie J M, et al. Analysis of nucleosome positioning in promoters of miRNA genes and protein-coding genes. Chinese Sci Bull, 2010, 55, doi: 10.1007/s11434-009-3730-2 论 文 《中国科学》杂志社 SCIENCE CHINA PRESS 专题: 生物信息学 miRNA 基因和编码基因启动子区核小体定位分析 刘宏德, 张德金, 谢建明, 袁志栋, 马昕, 卢志远, 龚乐君, 孙啸* 东南大学生物电子学国家重点实验室, 南京 210096 * 联系人, E-mail: xsun@https://www.360docs.net/doc/307809818.html, 2009-04-22 收稿, 2009-08-28 接受 国家自然科学基金资助项目(60671018, 30800209) 摘要 研究了把基因启动子区的核小体定位对于分析基因的转录调控具有重要意义. 利用核小体定位的预测技术——弯曲度谱, 分析了编码基因和miRNA 基因启动子周围核小体定位的特征. 基因的转录起始位点处, 有一个核小体缺失区域, 且在下游约200 bp 处, 有较强的核小体定位信号. 独立转录的内含子miRNA 基因与基因间区miRNA 基因, 在启动子区具有相似的核小体定位特征, 在上游0 ~ ?400 bp 间, 有一个较宽的核小体缺失区域, 在该区域分布有较多的转录因子结合位点; 而依赖编码基因转录的内含子miRNA 基因, 其启动子与蛋白编码基因启动子具有相似的核小体定位特征, 在转录起始位点上游?200 ~ ?400 bp 和?400 ~ ?600 bp 处, 各有一个较强的核小体定位. 这些结果表明, 独立转录的miRNA 基因(包括基因间区miRNA 和独立转录内含子miRNA)和蛋白编码基因, 在启动子区可能具有不同的核小体定位特征. 核小体定位不仅参与编码基因的转录调节, 也影响miRNA 基因的转录. 关键词 核小体定位 启动子 miRNA 基因 75%~90%的真核基因组DNA 包裹在组蛋白八联体上形成核小体[1]. 核小体定位是指DNA 双螺旋相对于组蛋白核的位置. 这种定位作用封闭了位于核小体上的蛋白结合位点[2,3], 进而阻止蛋白质与DNA 的结合, 以此达到调节基因转录的作用. 基因启动子结构和功能的分析对于研究转录调节、构建基因间相互作用网络等都具有重要意义. 目前, 对于编码基因启动子的研究较多, 认识较深入. miRNA 由于在细胞的发育分化、疾病(癌症)的发生发展中的有重要作用, 而备受关注 [4,5] , 但由于miRNA 前体(pri-RNA)的不稳定性和miRNA 的组织特异表达量低等原因, 使得对miRNA 基因自身的转录机制尚不明确[6]. 而从核小体定位的角度分析miRNA 基因的启动子将有助于理解miRNA 基因的转录调节机制[6,7]. 本文利用作者开发的核小体预测技术——弯曲度谱, 分别分析了编码基因、基因间miRNA 基因和内含子miRNA 基因的启动子区域核小体分布的特征. 结果显示独立转录的miRNA 基因与编码基因在启动子区核小体定位特征上有所差异. 这些分析对于理解miRNA 基因的转录过程具有重要作用. 1 数据和方法 (ⅰ) 数据. 用一段DNA 序列(人类20号染色体: 83.5~86.1 kb)来检验弯曲度谱的预测能力, 并将结果与Segal 等的模型结果进行比较(http://genie.weizm- ann.ac.il/soft-ware/nucleo_prediction.html, version 3.0)[8], 该段序列的核小体位置实验检测数据来自文献[9]. 672条蛋白编码基因启动子DNA 序列来自人类20号染色体, 序列取自UCSC (https://www.360docs.net/doc/307809818.html,/).
