限制性酶识别序列
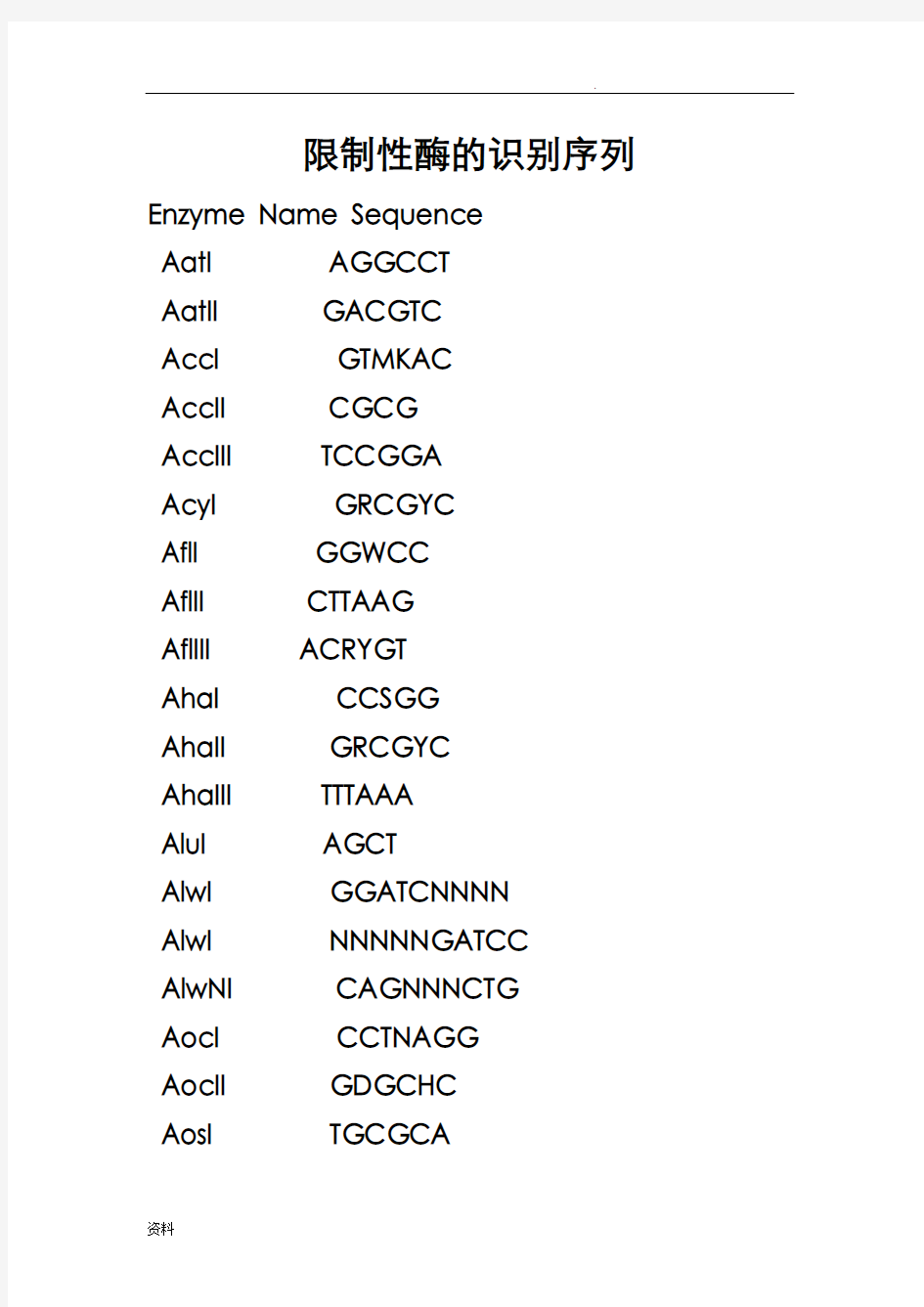

限制性酶的识别序列Enzyme Name Sequence
AatI AGGCCT
AatII GACGTC
AccI GTMKAC
AccII CGCG
AccIII TCCGGA
AcyI GRCGYC
AflI GGWCC
AflII CTTAAG
AflIII ACRYGT
AhaI CCSGG
AhaII GRCGYC
AhaIII TTTAAA
AluI AGCT
AlwI GGATCNNNN
AlwI NNNNNGATCC AlwNI CAGNNNCTG
AocI CCTNAGG
AocII GDGCHC
AosI TGCGCA
AosII GRCGYC ApaI GGGCCC ApaLI GTGCAC ApyI CCWGG AquI CYCGRG AseI ATTAAT
AspI GAANNNNTTC AspI GGTACC
AspI GACNNNGTC AsuI GGNCC AsuII TTCGAA
AvaI CYCGRG AvaII GGWCC AvaIII ATGCAT
AvrI CYCGRG AvrII CCTAGG AxyI CCTNAGG BalI TGGCCA BamHI GGATCC BanI GGYRCC BanII GRGCYC BanIII ATCGAT
BbeI GGCGCC
BbiII/AcyI GRCGYC
BbvI GCAGCNNNNNNNN
BbvI NNNNNNNNNNNNGCTGC BbvII GAAGACNN
BbvII NNNNNNGTCTTC
BcefI ACGGCNNNNNNNNNNNN BcefI NNNNNNNNNNNNNGCCGT BclI TGATCA
BcnI CCSGG
BglI GCCNNNNNGGC
BglII AGATCT
BinI NNNNNGATCC
BinI GGATCNNNN
BsePI GCGCGC
BsmAI GTCTC
BsmAI GAGAC
BsmI GAATGCN
BsmI GCATTC
BspI GDGCHC
BspHI TCATGA
BspMI ACCTGCNNNN
BspMI NNNNNNNNGCAGGT BspMII TCCGGA
BsrI ACTGGN
BsrI CCAGT
BssHII GCGCGC
BstBI TTCGAA
BstEII GGTNACC
BstI GGATCC
BstNI CCWGG
BstPI GGTNACC
BstUI CGCG
BstXI CCANNNNNNTGG BstYI RGATCY
BsuI CCTNAGG
CcrI CTCGAG
CfoI GCGC
CfrI RCCGGY
CfrI GGNCC
CfrI YGGCCR
ClaI ATCGAT
CviJI RGCY
CvnI CCTNAGG
DdeI CTNAG
DpnI GATC
DraI TTTAAA
DraII RGGNCCY DraIII CACNNNGTG DsaI CCRYGG
EaeI YGGCCR
EagI CGGCCG
EarI CTCTTC
EarI GAAGAG
EclXI CGGCCG
EcoI TACGTA
EcoI GGTCTCN
EcoI NNNNNGAGACC EcoI GGWCC
EcoIII AGCGCT
EcoI CGGCCG
EcoI CTGAAG
EcoI CTTCAG
EcoI CCTNAGG
EcoNI CCTNNNNNAGG EcoOI RGGNCCY
EcoRI GAATTC
EcoRII CCWGG
EcoRV GATATC
EcoTI CCWWGG
EcoTI ATGCAT
EcoTI GRGCYC
EheI GGCGCC
EspI GCTNAGC
FinI GTCCC
FinI GGGAC
FnuHI GCNGC
FokI GGATGNNNNNNNNN FokI NNNNNNNNNNNNNCATCC FspI TGCGCA
GdiII NNNNNYGGCCG
GdiII CGGCCRN
GsuI CTCCAG
GsuI CTGGAG
HaeI WGGCCW
HaeII RGCGCY
HaeIII GGCC
HapII CCGG
HgaI GACGCNNNNN
HgaI NNNNNNNNNNGCGTC HgiAI GWGCWC
HgiEII ACCNNNNNNGGT HhaI GCGC
HinI GRCGYC
HinPI GCGC
HincII GTYRAC
HindIII AAGCTT
HinfI GANTC
HpaI GTTAAC
HpaII CCGG
HphI GGTGANNNNNNNN HphI NNNNNNNTCACC KpnI GGTACC
KspI CTCTTCN
KspI NNNNGAAGAG
MaeI CTAG
MaeII ACGT
MaeIII GTNAC
MboI GATC
MboII GAAGANNNNNNNN
MboII NNNNNNNTCTTC MfeI CAATTG
MflI RGATCY
MluI ACGCGT
MmeI TCCRAC MmeI GTYGGA
MnlI CCTCNNNNNNN MnlI NNNNNNNGAGG MroI TCCGGA
MseI TTAA
MspI CCGG
MstI TGCGCA
MstII CCTNAGG
MvaI CCWGG
NaeI GCCGGC
NarI GGCGCC
NciI CCSGG
NcoI CCATGG
NdeI CATATG
NdeII GATC
NheI GCTAGC
NlaIII CATG
常用限制性内切酶酶切位点汇总
Acc65I识别位点AccI识别位点AciI识别位点AclI识别位点AcuI识别位点 AfeI识别位点AflII识别位点AflIII识别位点AgeI识别位点AhdI识别位点AleI识别位点AluI识别位点AlwI识别位点AlwNI识别位点ApaI识别位点ApaLI识别位点ApeKI识别位点ApoI识别位点AscI识别位点AseI识别位点AsiSI识别位点AvaI识别位点AvaII识别位点AvrII识别位点BaeI识别位点BamHI识别位点BanI识别位点BanII识别位点
BbvCI识别位点BbvI识别位点 BccI识别位点BceAI识别位点BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点BmgBI识别位点BmrI识别位点BmtI识别位点BpmI识别位点Bpu10I识别位点BpuEI识别位点BsaAI识别位点BsaBI识别位点BsaHI识别位点BsaI识别位点BsaJI识别位点BsaWI识别位点BsaXI识别位点BseRI识别位点BseYI识别位点
BsiEI 识别位点BsiHKAI 识别位点BsiWI识别位点BslI 识别位点BsmAI识别位点 BsmBI识别位点BsmFI识别位点BsmI识别位点BsoBI识别位点Bsp1286I识别位点BspCNI识别位点BspDI识别位点BspEI识别位点BspHI识别位点BspMI识别位点BspQI识别位点BsrBI识别位点BsrDI识别位点BsrFI识别位点BsrGI识别位点BsrI识别位点BssHII识别位点BssKI识别位点BssSI识别位点BstAPI识别位点BstBI识别位点BstEII识别位点BstNI识别位点
限制性内切酶酶切位点汇总
Acc65I识别位点 AccI识别位点 AciI识别位点 AclI识别位点 AcuI识别位点 AfeI识别位点 AflII识别位点 AflIII识别位点 AgeI识别位点 AhdI识别位点 AleI识别位点 AluI识别位点 AlwI识别位点 AlwNI识别位点 ApaI识别位点 ApaLI识别位点 ApeKI识别位点 ApoI识别位点 AscI识别位点 AseI识别位点 AsiSI识别位点 AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI识别位点 BanI识别位点 BanII识别位点
BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点 BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点 BmgBI识别位点 BmrI识别位点 BmtI识别位点 BpmI识别位点 Bpu10I识别位点 BpuEI识别位点 BsaAI识别位点 BsaBI识别位点 BsaHI识别位点 BsaI识别位点 BsaJI识别位点 BsaWI识别位点 BsaXI识别位点 BseRI识别位点 BseYI识别位点
BsiEI识别位点 BsiHKAI识别位点 BsiWI识别位点 BslI识别位点 BsmAI识别位点 BsmBI识别位点 BsmFI识别位点 BsmI识别位点 BsoBI识别位点 Bsp1286I识别位点 BspCNI识别位点BspDI识别位点 BspEI识别位点 BspHI识别位点 BspMI识别位点 BspQI识别位点 BsrBI识别位点 BsrDI识别位点 BsrFI识别位点 BsrGI识别位点 BsrI识别位点 BssHII识别位点 BssKI识别位点 BssSI识别位点 BstAPI识别位点 BstBI识别位点 BstEII识别位点 BstNI识别位点
常用限制性内切酶酶切位点保护残基
酶切位点保护碱基-PCR引物设计用于限制性内切酶 发布: 2010-05-24 20:19| 来源:生物吧| 编辑:刘浩| 查看: 161 次 本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,KpnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,SacII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI, 为什么要添加保护碱基? 在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。 其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。 该如何添加保护碱基? 添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。 添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。 为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。 实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。取1μg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。反应缓冲液含70mM Tris-HCl (pH 7.6), 10 mM MgCl2, 5 mMDTT及适量的NaCl或KCl(视酶的具体要求而定)。20%的PAGE(7M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。 本实验采用自连接的寡核苷酸作为对照。若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
限制性内切酶酶切位点_方便搜索
GACGTC CTGCAG Acc65I 识别位点 GTMKAC CAKMTG AccI 识别位点 GTMKAC CAKMTG AciI 识别位点 CCGC GGCG AclI 识别位点 AACGTT TTGCAA AcuI 识别位点 CTGAAG GACTTC AfeI 识别位点 AGCGCT TCGCGA CTTAAG GAATTC AflIII 识别位点 ACRYGT TGYRCA AgeI 识别位点 ACCGGT TGGCCA AhdI 识别位点 GACNNNNNGTC CTGNNNNNCAG AleI 识别位点 CACNNNNGTG GTGNNNNCAC AluI 识别位点 AGCT TCGA AlwI 识别位点 GGATC CCTAG
CAGNNNCTG GTCNNNGAC ApaI 识别位点 GGGCCC CCCGGG ApaLI 识别位点 GTGCAC CACGTG ApeKI 识别位点 GCWGC CGWCG ApoI 识别位点 RAATTY YTTAAR AscI 识别位点 GGCGCGCC CCGCGCGG AseI 识别位点 ATTAAT TAATTA GCGATCGC CGCTAGCG AvaI 识别位点 CYCGRG GRGCYC AvaII 识别位点 GGWCC CCWGG AvrII 识别位点 CCTAGG GGATCC BaeI 识别位点 NACNGTAYCN BamHI 识别位点 GGATCC CCTAGG BanI 识别位点 GGYRCC CCRYGG
2018年ecor1限制酶识别序列的特点是什么-范文模板 (2页)
2018年ecor1限制酶识别序列的特点是什么-范文模板 本文部分内容来自网络整理,本司不为其真实性负责,如有异议或侵权请及时联系,本司将立即删除! == 本文为word格式,下载后可方便编辑和修改! == ecor1限制酶识别序列的特点是什么 EcoRⅠ是从大肠杆菌R菌株中分离出来的第一个限制酶,下面是小编给大家整理的ecor1限制酶识别序列的特点,希望能帮到大家! ecor1限制酶识别序列的特点 EcoRⅠ切割位点: 5'-G AATTC-3' 3'-CTTAA G-5' 序列,切割位点在G与A之间,形成黏性末端。 限制酶的定义 限制性核酸内切酶是可以识别特定的核苷酸序列,并在每条链中特定部位的两个核苷酸之间的磷酸二酯键进行切割的一类酶,简称限制酶。根据限制酶的结构,辅因子的需求切位与作用方式,可将限制酶分为三种类型,分别是第一型(Type I)、第二型(Type II)及第三型(Type III)。Ⅰ型限制性内切酶既能催化宿主DNA的甲基化,又催化非甲基化的DNA的水解;而Ⅱ型限制性内切酶只催化非甲基化的DNA的水解。III型限制性内切酶同时具有修饰及认知切割的作用。 限制酶的由来 一般是以微生物属名的第一个字母和种名的前两个字母组成,第四个字母表示菌株(品系)。例如,从Bacillus amylolique faciens H中提取的限制性内切酶称为Bam H,在同一品系细菌中得到的识别不同碱基顺序的几种不同特异性的酶,可以编成不同的号,如HindⅡ、HindⅢ,HpaI、HpaⅡ,MboI、MboⅡ等。 别名:Endodeoxyribonuclease简称限制酶 酶反应限制性内切酶能分裂DNA分子在一限定数目的专一部位上。它能识别外源DNA并将其降解。
常用限制性内切酶酶切位点
AatII 识别位点 Acc65I 识别位点 AccI 识别位点 AciI 识别位点 AclI 识别位点 AcuI 识别位点 AfeI 识别位点 AflII 识别位点 AflIII 识别位点 AgeI 识别位点 AhdI 识别位点 AleI 识别位点 AluI 识别位点 AlwI 识别位点 AlwNI 识别位点 ApaI 识别位点 ApaLI 识别位点 ApeKI 识别位点 ApoI 识别位点 AscI 识别位点 AseI 识别位点 AsiSI 识别位点
AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI 识别位点 BanI识别位点 BanII识别位点 BbsI识别位点 BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点BcgI识别位点BciVI识别位点BclI识别位点 BfaI识别位点BfuAI识别位点BglI识别位点BglII识别位点BlpI识别位点Bme1580I识别位点BmgBI识别位点BmrI识别位点
BmtI 识别位点 BpmI 识别位点 Bpu10I 识别位点 BpuEI 识别位点 BsaAI 识别位点 BsaBI 识别位点 BsaHI 识别位点 BsaI 识别位点 BsaJI 识别位点 BsaWI 识别位点 BsaXI 识别位点 BseRI 识别位点 BseYI 识别位点 BsgI 识别位点 BsiEI 识别位点 BsiHKAI 识别位点 BsiWI 识别位点 BslI 识别位点 BsmAI 识别位点 BsmBI 识别位点 BsmFI 识别位点 BsmI 识别位点
酶切位点序列
ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列: 5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3'
酶Ⅰ的识别序列和切点是—G
2008.10.31 龙冈中学2008/2009学年度基因工程单元检测 高二生物(选修) 命题人:侍东升 班级:____________学号:__________姓名:_____________ 一、单项选择题:本部分包括20题,每题2分,共计40分。每题只有一个 ....选项最符合题意。 1、基因工程技术也称为DNA重组技术,其实施必须具备的四个必要条件是( ) A.目的基因限制酶运载体受体细胞 B.重组DNA RNA聚合酶限制酶连接酶 C.工具酶目的基因运载体受体细胞 D.模板DNA mRNA 质粒受体细胞 2、在DNA测序工作中,需要将某些限制性核酸内切酶的限制位点在DNA上定位,使其成为DNA 分子中的物理参照点,这项工作叫做“限制酶图谱的构建”。假设有以下一项实验:用限制酶HindⅢ、BamHI和二者的混合物分别降解一个4 kb(1 kb即1千个碱基对)大小的线性DNA 分子,降解产物分别进行凝胶电泳,在电场的作用下,降解产物分开,如下图所示。据此分析,这两种限制性核酸内切酶在该DNA分子上的限制位点数目是 ( ) A.H ind Ⅲ 1个,BamHI 2个 B.Hind Ⅲ 2个,BamHI 3个 C.Hind Ⅲ 2个,BamHI 1个 D.Hind Ⅲ和BamHI各有2个 3、水母发光蛋白由236个氨基酸构成,其中Asp、Gly、Ser构成发光环,现已将这种蛋白质 的基因作为生物转基因的标记,在转基因技术中,这种蛋白质的作用是( ) A、促使目的基因导入宿主细胞中B、使目的基因容易被检测出来 C、促使目的基因在宿主细胞中复制 D、使目的基因容易成功表达 4、碱基互补配对发生在下列哪些生理过程或生物技术中( ) ①种子的萌发②病毒的增殖过程③细菌的二分裂过程④目的基因与运载体的结合 ⑤DNA探针的使用⑥分泌蛋白的加工和运输 A.①②③④⑤B.①②③④⑤⑥C.②④⑤D.②③⑤⑥5、下列有关基因工程技术的正确叙述是 ( ) A.重组DNA技术所用的工具酶是限制酶、连接酶和运载体 B.所有的限制酶都只能识别同一种特定的核苷酸序列 C.选用细菌作为重组质粒的受体细胞是因为细菌繁殖快 D.只要目的基因进入了受体细胞就能成功实现表达 6、人的糖蛋白必须经内质网和高尔基体进一步加工合成,通过转基因技术,可以使人的糖蛋 白基因得以表达的受体细胞是( ) A、大肠杆菌 B、T4噬菌体 C、酵母菌 D、质粒DNA 7、下列各项与蛋白质结构多样性无关的是( ) A、氨基酸的种类、数目、排列顺序 B、构成蛋白质的多肽链的数目 C、构成蛋白质的肽链的空间结构 D、氨基酸至少含一个氨基和一个羧基 8、在已知序列信息的情况下,获取目的基因的最方便方法是( ) A、化学合成法 B、基因组文库法 C、C DNA文库法 D、聚合酶链反应 9、有关基因工程的叙述正确的是( ) A.限制性内切酶只在获得目的基因时才用 B.重组质粒的形成在细胞内完成 C.质粒都可作运载体 D.蛋白质的结构可为合成目的基因提供资料 10、基因工程常用的受体细胞有( ) ①大肠杆菌②枯草杆菌③支原体④动植物细胞 A.①②③④ B.①②③ C.②③④D.①②④ 11、构建基因组DNA文库时,首先要分离细胞的( ) A、染色体DNA B、线粒体DNA C、总mRNA D、tRNA 12、就分子结构而论,质粒是( ) A、环状双链DNA分子 B、环状单链DNA分于 C、环状单链RNA分子 D、线状双链DNA分子 13、若利用基因工程技术培育能固氮的水稻新品种,其在环保上的重要意义是( ) A.减少氮肥的使用量,降低生产成本 B.减少氮肥的使用量,节约能源 C.避免氮肥过多引起环境污染 D.改良土壤结构
史上最全限制性内切酶酶切位点汇总
A系列 AatII识别位点 Acc65I识别位点 AccI识别位点 AciI识别位点 AclI识别位点 AcuI识别位点 AfeI识别位点 AflII识别位点 AflIII识别位点 AgeI识别位点 AhdI识别位点 AleI识别位点 AluI识别位点 AlwI识别位点 AlwNI识别位点 ApaI识别位点 ApaLI识别位点 ApeKI识别位点 ApoI识别位点 AscI识别位点 AseI识别位点 AsiSI识别位点 AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI识别位点 BanI识别位点 BanII识别位点
BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点 BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点 BmgBI识别位点 BmrI识别位点 BmtI识别位点 BpmI识别位点 Bpu10I识别位点 BpuEI识别位点 BsaAI识别位点 BsaBI识别位点 BsaHI识别位点 BsaI识别位点 BsaJI识别位点 BsaWI识别位点 BsaXI识别位点 BseRI识别位点 BseYI识别位点
BsiEI识别位点 BsiHKAI识别位点 BsiWI识别位点 BslI识别位点 BsmAI识别位点 BsmBI识别位点 BsmFI识别位点 BsmI识别位点 BsoBI识别位点 Bsp1286I识别位点 BspCNI识别位点BspDI识别位点 BspEI识别位点 BspHI识别位点 BspMI识别位点 BspQI识别位点 BsrBI识别位点 BsrDI识别位点 BsrFI识别位点 BsrGI识别位点 BsrI识别位点 BssHII识别位点 BssKI识别位点 BssSI识别位点 BstAPI识别位点 BstBI识别位点 BstEII识别位点 BstNI识别位点
限制酶识别序列
二轮小专题 限制酶识别序列 一.回文诗: 静思伊久阻归期, 久阻归期忆别离; 忆别离时闻漏转, 时闻漏转静思伊。 赏花归去马如飞, 去马如飞酒力微。 酒力微醒时已暮, 醒时已暮赏花归。 二.DNA回文序列 限制性内切酶Ⅱ类酶有EcoR I、BamH I、Hind Ⅱ、Hind Ⅲ等。其分子量小于105道尔顿;反应只需Mg2+;最重要的是在所识别的特定碱基顺序上有特异性的切点,因而DNA分子经过Ⅱ类酶作用后,可产生特异性的酶解片断,这些片断可用凝胶电泳法进行分离、鉴别。限制性内切酶识别DNA序列中的回文序列。有些酶的切割位点在回文的一侧(如EcoR I、BamH I、Hind等),因而可形成粘性末端,另一些Ⅱ类酶如Alu I、BsuR I、Bal I、Hal Ⅲ、HPa I、Sma I等,切割位点在回文序列中间,形成平整末端。Alu I的切割位点如下:5'-A G^C T-3' 3'-T C^G A-5'
酶类型识别序列 ApaI Type II restriction enzyme 5'GGGCC^C 3' BamHI Type II restriction enzyme 5' G^GATCC 3' BglII Type II restriction enzyme 5' A^GATCT 3' EcoRI Type II restriction enzyme 5' G^AATTC 3' HindIII Type II restriction enzyme 5' A^AGCTT 3' KpnI Type II restriction enzyme 5' GGTAC^C 3' NcoI Type II restriction enzyme 5' C^CATGG 3' NdeI Type II restriction enzyme 5' CA^TATG 3' NheI Type II restriction enzyme 5' G^CTAGC 3' NotI Type II restriction enzyme 5' GC^GGCCGC 3' SacI Type II restriction enzyme 5' GAGCT^C 3' SalI Type II restriction enzyme 5' G^TCGAC 3' SphI Type II restriction enzyme 5' GCATG^C 3' XbaI Type II restriction enzyme 5' T^CTAGA 3' XhoI Type II restriction enzyme 5' C^TCGAG 3'
常用限制性内切酶酶切位点总结
常用限制性内切酶酶切位点总结
————————————————————————————————作者:————————————————————————————————日期:
Acc65I识别位点 AccI识别位点 AciI识别位点 AclI识别位点 AcuI识别位点 AfeI识别位点 AflII识别位点 AflIII识别位点 AgeI识别位点 AhdI识别位点 AleI识别位点 AluI识别位点 AlwI识别位点 AlwNI识别位点 ApaI识别位点 ApaLI识别位点 ApeKI识别位点 ApoI识别位点 AscI识别位点 AseI识别位点 AsiSI识别位点 AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI识别位点 BanI识别位点 BanII识别位点
BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点 BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点 BmgBI识别位点 BmrI识别位点 BmtI识别位点 BpmI识别位点 Bpu10I识别位点 BpuEI识别位点 BsaAI识别位点 BsaBI识别位点 BsaHI识别位点 BsaI识别位点 BsaJI识别位点 BsaWI识别位点 BsaXI识别位点 BseRI识别位点 BseYI识别位点
BsiEI识别位点 BsiHKAI识别位点 BsiWI识别位点 BslI识别位点 BsmAI识别位点 BsmBI识别位点 BsmFI识别位点 BsmI识别位点 BsoBI识别位点 Bsp1286I识别位点 BspCNI识别位点BspDI识别位点 BspEI识别位点 BspHI识别位点 BspMI识别位点 BspQI识别位点 BsrBI识别位点 BsrDI识别位点 BsrFI识别位点 BsrGI识别位点 BsrI识别位点 BssHII识别位点 BssKI识别位点 BssSI识别位点 BstAPI识别位点 BstBI识别位点 BstEII识别位点 BstNI识别位点
限制性内切酶酶切位点汇总
限制性内切酶酶切位点汇AatII识别位点 Acc65I识别位点AccI识别位点AciI识别位点AclI识别位点AcuI识别位点AfeI识别位点AflII识别位点AflIII识别位点AgeI识别位点AhdI识别位点AleI识别位点AluI识别位点AlwI识别位点AlwNI识别位点 ApaI识别位点 ApaLI识别位点 ApeKI识别位点 ApoI识别位点 AscI识别位点 AseI识别位点 AsiSI识别位点 AvaI识别位点 AvaII识别位点 AvrII识别位点 BaeI识别位点 BamHI识别位点 BanI识别位点 BanII识别位点 BbsI识别位点 BbvCI识别位点 BbvI识别位点 BccI识别位点 BceAI识别位点 BcgI识别位点 BciVI识别位点 BclI识别位点 BfaI识别位点 BfuAI识别位点 BglI识别位点 BglII识别位点
BlpI识别位点 Bme1580I识别位点BmgBI识别位点BmrI识别位点 BmtI识别位点BpmI识别位点 Bpu10I识别位点BpuEI识别位点BsaAI识别位点BsaBI识别位点BsaHI识别位点BsaI识别位点BsaJI识别位点BsaWI识别位点BsaXI识别位点BseRI识别位点BseYI识别位点 BsgI识别位点 BsiEI识别位点 BsiHKAI识别位点 BsiWI识别位点 BslI识别位点 BsmAI识别位点 BsmBI识别位点 BsmFI识别位点 BsmI识别位点 BsoBI识别位点 Bsp1286I识别位点 BspCNI识别位点 BspDI识别位点 BspEI识别位点 BspHI识别位点 BspMI识别位点 BspQI识别位点 BsrBI识别位点 BsrDI识别位点 BsrFI识别位点 BsrGI识别位点 BsrI识别位点 BssHII识别位点 BssKI识别位点 BssSI识别位点 BstAPI识别位点 BstBI识别位点 BstEII识别位点 BstNI识别位点 BstUI识别位点 BstXI识别位点
常用限制性内切酶酶切位点汇总
ApaI识别位点Acc65I识别位点 ApaLI识别位点AccI识别位点 ApeKI识别位点AciI识别位点 ApoI识别位点AclI识别位点 AscI识别位点AcuI识别位点 AseI识别位点AfeI识别位点 AsiSI识别位点AflII识别位点 AvaI识别位点AflIII识别位点 AvaII识别位点AgeI识别位点 AvrII识别位点AhdI识别位点 BaeI识别位点AleI识别位点 BamHI识别位点AluI识别位点 BanI识别位点AlwI识别位点 BanII识别位点AlwNI识别位点
BmrI识别位点BbvCI识别位点 BmtI识别位点BbvI识别位点 BpmI识别位点BccI识别位点 Bpu10I识别位点BceAI识别位点 BpuEI识别位点BcgI识别位点 BsaAI识别位点BciVI识别位点 BsaBI识别位点BclI识别位点 BsaHI识别位点BfaI识别位点 BsaI识别位点BfuAI识别位点 BsaJI识别位点BglI识别位点 BsaWI识别位点BglII识别位点 BsaXI识别位点BlpI识别位点 BseRI识别位点Bme1580I识别位点 BseYI识别位点BmgBI识别位点
BspMI识别位点BsiEI识别位点 BspQI识别位点BsiHKAI识别位点 BsrBI识别位点BsiWI识别位点 BsrDI识别位点BslI识别位点 BsrFI识别位点BsmAI识别位点 BsrGI识别位点BsmBI识别位点 BsrI识别位点BsmFI识别位点 BssHII识别位点BsmI识别位点 BssKI识别位点BsoBI识别位点 BssSI识别位点Bsp1286I识别位点 BstAPI识别位点BspCNI识别位点 BstBI识别位点BspDI识别位点 BstEII识别位点BspEI识别位点 BstNI识别位点BspHI识别位点
常见限制性内切酶识别序列(酶切位点)
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine. ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3' 当然,上面总结的这些肯定不全,要查找更多内切酶的识别序列,你还可以选择下面几种方法: 1. 查你所使用的内切酶的公司的目录或者网站;NEB网站上提供的识别序列图表下载 2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;
限制性酶切常见问题分析
限制性内切酶酶切注意事项 在进行限制性酶切反应过程中,影响限制性酶切反应效果的因素较多,要设计酶切反应,一般均应考虑诸如酶切系统、温度条件、反应体积、底物性质及星型反应等因素。根据各种不同的 条件,酶切反应的设计一般应注意以下问题: 1. 大多数限制酶贮存在50%甘油溶液中,以避免在-20℃条件下结冰。当最终反应液中甘油浓度大于12%时,某些限制酶的识别特异性降低,从而产生星活性,更高浓度的甘油会抑制酶活 性。因此加入反应的酶体积不超过反应总体积的1/10,避免限制酶活性受到甘油的影响。 2. 浓缩的限制酶可在使用前用1×限制酶缓冲液稀释,但切勿用水稀释以免酶失活,用水稀释的酶不能长期保存。 3. 反应体系中Mg2+是限制酶仅有的共同因子,当用其它二价阳离子代替Mg2+或加入能螯合Mg2+的EGTA或EDTA时,酶活性会被抑制,或改变酶的特异性,导致星型反应。 4. 多种因素可引发星型反应:①非最适的pH;②Co2+、Mn2+、2n2+取代Mg2+;③酶浓度大于25u/ug;④盐浓度降低;⑤高浓度甘油的存在(>12%);⑥有机溶剂的存在。 5. 反应混合物中DNA底物的浓度不宜太大,小体积中过高浓度的DNA会形成粘性DNA溶液抑制酶的扩散,并降低酶活性。建议酶切反应的DNA浓度为0.1-0.4ug/ul。 6. 酶切反应所加入的酶量应适中,根据底物的种类、量的多少和体积的大小而定,对不同的限制酶,各厂家均有一最大的消化量指标可参考。 7. 当要用两种或两种以上限制酶切割DNA时,如果这些酶可以在同种缓冲液中作用良好,则两种酶可同时切割,如果这些酶所要求的缓冲液有所不同,则可采用以下两种替代方法:①先用在低离子强度的缓冲液中活性高的酶切割DNA,然后加入适量NaCl及第二种酶,继续反应; ②使用能够使多数内切酶均表现较高活性的单种缓冲液。 8.用同一种酶切割不同的DNA时,所需酶量不同,可根据DNA底物上酶切位点的多少与 λDNA存在位点的数目比较后,决定用酶量。对于超螺旋DNA,基因组DNA、琼脂糖包埋的DNA,使用的酶单位活性应适当加大。 9. 反应混合液中加入浓度为0.1mg/ml的BSA,可维持酶的稳定性。 10.酶切底物DNA应具备一定的纯度,其溶液中不能含有迹量酚、氯仿、乙醚,大于10mM 的EDTA,去污剂SDS以及过量的盐离子浓度,否则会不同程度地影响限制酶的活性。 11.D NA碱基上的甲基化修饰也是影响酶切的一个重要因素,所以转化实验所选择的受体菌株应考虑到使用的菌株中的酶修饰系统。 12.要保证酶作用时的最佳反应条件(ph, 温度)和底物用量,酶反应才能有效地进行。 13.反应取酶时应使用无菌吸头,以免污染酶液,同时应尽量缩短酶在温室的放置时间。 14.高于37℃或需长时间保温时,可加入矿物油覆盖在反应液上以减少水分蒸发。 15.反应混合物混匀时,应避免强烈振荡以保证不使内切酶变性及DNA大分子的完整。 16.反应前的低速离心是必要的,这可使因混匀吸附于管壁上的液滴全部沉至管底。 17.用已硅化的离心管进行酶切反应,可获得更佳的酶切效果,否则DNA可能粘附于管壁而影响酶切效果。 18.终止酶反应可根据需要采用不同的方法:①酶切后不需进行下一步反应,可加入含EDTA 的终止液终止反应;②若需进一步反应(如连接,切割等),可将反应管置65℃保温20-30分钟,以灭活酶终止反应。③可用酚/氯仿抽提,乙醇沉淀获得较纯DNA进行下一步酶学操作。
常见限制性内切酶识别序列
常见限制性内切酶识别序列(酶切位点)(BamHI、EcoRI、HindIII、NdeI、 XhoI等) Time:2009-10-22 PM 15:38Author:bioer Hits: 7681 times 在分子克隆实验中,限制性内切酶是必不可少的工具酶。 无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T 载就不必考虑了)。先将引物设计好,然后添加酶切识别序列到引物5' 端。常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。 下面是一些常用的II型内切酶的识别序列,仅供参考。先介绍一下什么是II型内切酶吧。 The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine. 酶类型识别序列 ApaI Type II restriction enzyme 5'GGGCC^C 3' BamHI Type II restriction enzyme 5' G^GATCC 3' BglII Type II restriction enzyme 5' A^GATCT 3' EcoRI Type II restriction enzyme 5' G^AATTC 3' HindIII Type II restriction enzyme 5' A^AGCTT 3'
酶的使用说明
限制酶使用说明 一、分类 目前,已被发现的限制酶,根据其反应的必须因子和切断点等特性,被分为以下三大类:类别反应必须因子切点酶例 I 型 s-腺苷基蛋氨酸、ATP、Mg2+识别部位和切点不同,切断部位不定Eco B、Eco K II 型 Mg2+切断识别部位或其附近的特定部位Eco R I、Bam H I III 型 ATP、Mg2+识别部位和切点不同,但切断特定部位Eco P I、Hin f III 应用于基因工程研究用的限制酶,一般全是II型酶,现在市场上销售的酶都属于II型酶, 这些限制酶由于其反应条件和底物DNA种类的不同,其切断状况及出现Star活性的频率等各有不同,并且其程度也根据酶的不同而千差万别。因而在使用限制酶时,必须对这些要素充分注意,确保目标序列的切断反应能顺利进行,下面具体介绍一下使用限制酶时的一些注意点。 二、注意事项 1. 甲基化的影响 从带有DNA甲基化酶基因的宿主菌中制备的DNA,其碱基的一部分已经被甲基化,因此即便使用能够识别、切断被甲基化部分的序列的限制酶,也几乎无法切断被甲基化的部分。被甲基化的部位,根据底物DNA 及宿主种类的不同而不同。例如宿主菌为大肠杆菌的情况下,根据宿主的种类有以下两种情况: 在进行转化时,通常使用的菌株为C600、HB101、JM109等,因为都带有dam、dcm甲基化酶,所以使用这些菌株制备的DNA时,必须注意。另外,动物由来的DNA,CG序列多为5m CG;植物由来的DNA,CG 及CNG序列多为5m CG和5m CNG。 2. Star活性 限制酶在一些特定条件下使用时,对于底物DNA的特异性可能降低。即可以把与原来识别的特定的DNA 序列不同的碱基序列切断,这个现象叫Star活性。Star活性出现的频率,根据酶、底物DNA、反应条件的不同而不同,可以说几乎所有的限制酶都具有Star活性。并且,它们除了识别序列的范围增大之外,还发现了在DNA的一条链上加入切口的单链切口活性,所以为了极力抑制Star活性,一般情况下,即使会降低反应性能,我们也提倡在低甘油浓度、中性pH、高盐浓度条件下进行反应。
常用限制性内切酶酶切位点汇总
Acc65I识别位点AccI识别位点AciI 识别位点AclI识别位点AcuI 识别位点AfeI 识别位点AflII 识别位点AflIII 识别位点AgeI 识别位点AhdI 识别位点AleI识别位点AluI识别位点AlwI识别位点AlwNI识别位点ApaI识别位点ApaLI识别位点ApeKI识别位点ApoI识别位点AscI识别位点AseI识别位点AsiSI识别位点AvaI识别位点AvaII识别位点AvrII识别位点BaeI识别位点BamHI识别位点BanI识别位点BanII识别位点
BbvCI识别位点BbvI识别位点 BccI 识别位点BceAI识别位点BcgI 识别位点BciVI 识别位点 BclI 识别位点 BfaI 识别位点 BfuAI 识别位点 BglI 识别位点 BglII识别位点 BlpI识别位点 Bme1580I识别位点BmgBI识别位点BmrI识别位点BmtI识别位点BpmI识别位点Bpu10I识别位点BpuEI识别位点BsaAI识别位点BsaBI识别位点BsaHI识别位点BsaI识别位点BsaJI识别位点BsaWI识别位点BsaXI识别位点BseRI识别位点BseYI识别位点
BsiEI识别位点BsiHKAI识别位点BsiWI识别位点BslI识别位点BsmAI识别位点BsmBI识别位点BsmFI 识别位点BsmI识别位点BsoBI识别位点Bsp1286I识别位点BspCNI识别位点BspDI识别位点BspEI识别位点BspHI识别位点BspMI识别位点BspQI识别位点BsrBI识别位点BsrDI识别位点BsrFI识别位点BsrGI识别位点BsrI识别位点BssHII识别位点BssKI识别位点BssSI识别位点BstAPI识别位点BstBI识别位点BstEII识别位点BstNI识别位点
常用的内切酶的识别序列
在分子克隆实验中,限制性内切酶是必不可少的工具酶。 无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T载就不必考虑了)。先将引物设计好,然后添加酶切识别序列到引物5’端(英语怎么说?)。常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。 下面,我就总结了一些常用的内切酶的识别序列,仅供各位参考。下面这些内切酶都属于II型内切酶。 先介绍一下什么是II型内切酶吧。 The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine. ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3' 当然,上面总结的这些肯定不全,要查找更多内切酶的识别序列,你还可以选择下面几种方法: 1. 查你所使用的内切酶的公司的目录或者网站;NEB网站上提供的识别序列图表下载 2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息; 3. 推荐到NEB的REBASE数据库去查(网址:https://www.360docs.net/doc/b810052767.html,/rebase/rebase.html) 当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考: NEB公司网站提供的保护碱基参考表下载
