mRNA-lncRNA-甲基化联合分析
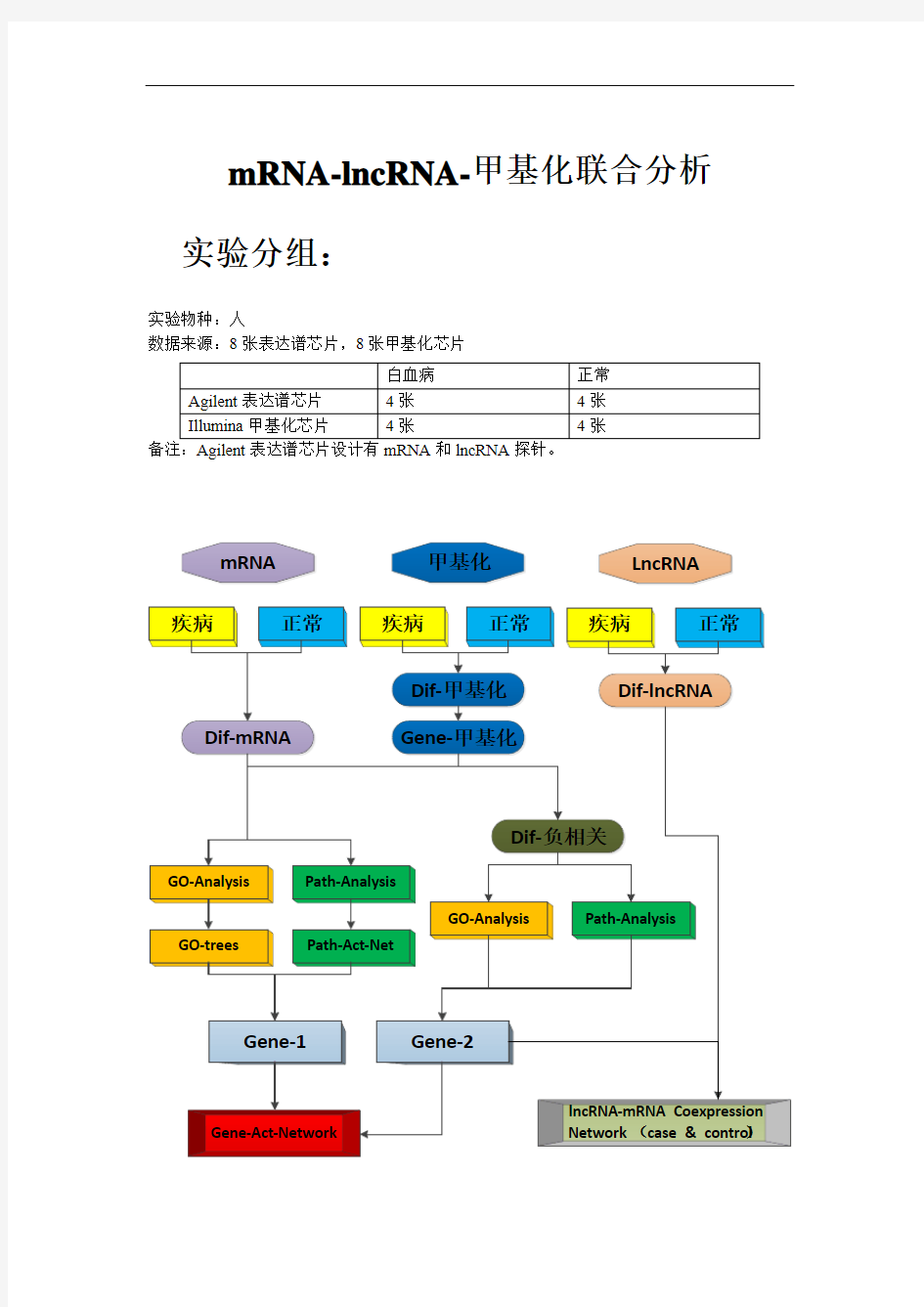
mRNA-lncRNA-甲基化联合分析实验分组:
实验物种:人
数据来源:8张表达谱芯片,8张甲基化芯片
备注:Agilent表达谱芯片设计有mRNA和lncRNA探针。
疾病mRNA
正常
Dif-mRNA 疾病
LncRNA
正常Dif-lncRNA
GO-Analysis Path-Analysis GO-trees Path-Act-Net
Gene-1
Gene-Act-Network
lncRNA-mRNA Coexpression
Network (case & control)疾病
甲基化
正常
Dif-甲基化
Gene-甲基化
Dif-负相关
GO-Analysis Path-Analysis
Gene-2
白血病正常
Agilent表达谱芯片4张4张Illumina甲基化芯片4张4张
DNA甲基化测序服务DNA甲基化是表观遗传学(Epigen
DNA甲基化测序服务 DNA甲基化是表观遗传学(Epigenetics)的重要组成部分,在维持正常细胞功能、遗传印记、胚胎发育以及人类肿瘤发生中起着重要的作用,是目前新的研究热点之一。在哺乳动物中,甲基化一般发生在CpG的胞嘧啶5位碳原子上,通过使用5’-甲基胞嘧啶抗体富集高甲基化的DNA片段,并结合Illumina高通量测序平台,对所有富集的DNA片段进行高通量测序,研究人员能够获得全基因组范围内高精度的甲基化状态,为深入的表观遗传调控分析提供了更有利的切入点。 DNA甲基化测序技术优势 ?灵活度高:能够直接对任意物种的高甲基化片段进行测序,无需已知的基因组序列信息。 ?检测范围广:覆盖整个基因组范围的甲基化区域。 ?精确度高:能够在实际结合位点50个碱基范围内精确定位。 ?数字化信号:直接对甲基化片段进行测序和定量,不存在传统芯片杂交的荧光模拟信号带来的交叉反应和背景噪音问题。 图1 MeDIP-Seq测序技术能够覆盖整个基因组区域包括启动子区、内含子、外显子、基因间,甚至是一些重复序列,并能分析这些区域各自的甲基化程度,右上角的不同颜色就代表了不同的甲基化程度(摘自Down TA., et al, 2008, Nature Biotechnology)。
康成生物提供的DNA甲基化测序服务流程: 1. 甲基化DNA免疫共沉淀(MeDIP) 2. 测序文库构建 ?双链DNA末端修复及3’末端加’A’ ?使用特定的测序接头连接DNA片段两端 ?高保真聚合酶扩增构建的测序文库 3. DNA成簇(Cluster)扩增 4. 高通量测序(Illumina Genome Analyzer IIx) 5. 数据分析 ?原始数据读取 ?与数据库比对并进行注释 ?确定甲基化位点 ?深层次数据分析 6. 提供实验报告 ?原始数据报告(Fasta-Q格式),包含所有 测序序列信息,碱基读取质量评估 ?基本数据分析报告(Excel表格),包含有 效序列的序列信息、与参考基因组比对后的 注释信息等。 ?高级数据分析(应客户要求定制),如甲基化区域(enriched region)鉴定,分析两样本间甲基化水平有差异的区域并注释,结合表达谱分析其对基因的表达调控。
DNA甲基化分析方法
· 489 · 《生命的化学》2008年28卷4期CHEMISTRY OF LIFE 2008,28(4) ● 技术与方法 文章编号: 1000-1336(2008)04-0489-03 DNA甲基化分析方法 肖正中 邬苏晓 (韶关学院英东生物工程学院,韶关 512005) 摘要:DNA甲基化是表观遗传学的重要研究内容之一。甲基化分析的方法多且研究难度大,各种方法都有其一定的优势和不足。本文综述了基因组DNA甲基化和特定DNA片段甲基化状态分析方法新进展,为研究者提供参考。关键词:DNA甲基化;CpG岛;表观遗传学中图分类号:Q334 收稿日期:2008-04-12 作者简介:肖正中(1972-),男,博士,讲师,E-mail:xzzwsx@sina.com;邬苏晓(1972-),女,硕士,讲师,联系作者,E-mail:sxwa4035@sina.com DNA甲基化是表观遗传学的重要研究内容之一,它可以在转录水平抑制基因的表达。甲基化通常发生在胞嘧啶的C5位,形成5-甲基胞嘧啶(5mC),甲基化的胞嘧啶多位于CpG岛上。CpG岛是CpG二联核苷富集区域,CG含量大于50%,长约200 ̄500 bp。哺乳动物DNA甲基化的模式只有5mC这一形式,真核生物中大约2% ̄7%的胞嘧啶被甲基化修饰。1. 基因组DNA甲基化分析方法 早期的基因组DNA甲基化分析技术,如SssI甲基转移酶分析法、氯乙醛反应法、免疫学抗体技术等,已不能满足现代表观遗传学研究的需求。近年来常用的基因组甲基化分析方法有以下两种。1.1 甲基化敏感扩增多态性技术 甲基化敏感扩增多态性(methylation sensitive amplification polymorphism,MSAP)技术由Reyna-lópez等报道,并被用于检测双相型真菌的DNA甲基化[1] ,它 是在扩增片段长度多 态性(amplified fragment length polymorphism, AFLP)技术的基础上建立起来的。其基本程序是:提取高质量基因组DNA,分别用EcoRI/HpaII,EcoRI/MspI两组酶组合对基因组DNA进行双酶切,并连上相应的限制性内切酶的接头,然后以接头序列设计的预扩增引物,进行PCR扩增。扩增产物稀释后,再加入带有选择性碱基的引物,进行第二次PCR扩增,扩 增产物变性后在6%的序列胶上电泳,最后采用银染或同位素放射自显影方法处理序列胶,统计和分析DNA条带。这种方法在研究动植物基因组甲基化上有广泛应用[2-6]。 MSAP技术相对其他测定DNA甲基化程度的技术有如下优点:(1)不需知道被测DNA的序列信息,在不同生物上具有通用性,可用于DNA序列背景知识未知的生物。(2)操作相对简便,在AFLP技术体系的基础无需改进,即可操作。(3)可在全基因组范围检测CCGG位点的胞嘧啶甲基化变化。MSAP技术的局限性在于不能完成非CCGG位点的胞嘧啶甲基化。1.2 高效液相层析及相关方法 高效液相层析(highperformance liquid chromatography, HPLC)能够定量测定基因组整体甲基化水平,其过程是:先将DNA样品经盐酸或氢氟酸水解成碱基,水解产物通过色谱柱,将结果与标准品比较,紫外光测定吸收峰值,计算5mC/(5mC+5C)的积分面积得出基因组整体的甲基化水平。Fraga等[7]运用高效毛细管电泳法(high per-formance capillary electrophoresis, HPCE)处理DNA水解产物确定5mC的水平,相比HPLC,HPCE更简便、快速、经济。HPLC及HPCE测定基因组整体DNA甲基化水平的敏感性均较高。2. 特定DNA片段甲基化检测方法 2.1 亚硫酸氢盐预处理法 (1)甲基化特异性PCR。甲基化特异性PCR(MSP)是Herman等[8]首先提出的一种 检测基因组DNA甲基化水平的常用方法。该法是将DNA经亚硫酸氢钠处理,非甲基化的胞嘧啶转变为尿嘧啶,而甲基化的胞嘧啶保持不变。在PCR反应
高通量测序常用名词汇总
高通量测序常用名词汇总 技术支持 Q20值是指的测序过程碱基识别(Base Calling)过程中,对所识别的碱基给出的错误概率. 如果质量值是Q20,则错误识别的概率是1%,即错误率1%,或者正确率是99%; 如果质量值是Q30,则错误识别的概率是0.1%,即错误率0.1%,或者正确率是99.9%; 如果质量值是Q40,则错误识别的概率是0.01%,即错误率0.01%,或者正确率是99.99%; 你发现规律没有,Q“N”0的质量值,就是正确率有N个9的百分比,这样就非常容易记忆了. 基因高通量测序中,每测一个碱基会给出一个相应的质量值,这个质量值是衡量测序准确度的。碱基的质量值13,错误率为5%,20的错误率为1%,30的错误率为0.1%。行业中Q20与Q30则表示质量值≧20或30的碱基所占百分比。例如一共测了1G的数据量,其中有0.9G的碱基质量值大于或等于20,那么Q20则为90%。Q20值是指的测序过程碱基识别(Base Calling)过程中,对所识别的碱基给出的错误概率。质量值是Q20,则错误识别的概率是1%,即错误率1%,或者正确率是99%; 质量值是Q30,则错误识别的概率是0.1%,即错误率0.1%,或者正确率是99.9%; 质量值是Q40,则错误识别的概率是0.01%,即错误率0.01%,或者正确率是99.99%; 一代测序技术:即传统的Sanger测序法,Sanger法是根据核苷酸在待定序列模板上的引物点开始,随机在某一个特定的碱基处终止,并且在每个碱基后面进行荧光标记,产生以A、T、C、G结束的四组不同长度的一系列核苷酸,每一次序列测定由一套四个单独的反应构成,每个反应含有所有四种脱氧核苷酸三磷酸(dNTP),并混入限量的一种不同的双脱氧核苷三磷酸(ddNTP)。由于ddNTP缺乏延伸所需要的3-OH 基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止,使反应得到一组长几百至几千碱基的链终止产物。它们具有共同的起始点,但终止在不同的的核苷酸上,可通过高分辨率变性凝胶电泳分离大小不同的片段,通过检测得到DNA碱基序列。 二代测序技术:next generation sequencing(NGS)又称为高通量测序技术,与传统测序相比,二代测序技术可以一次对几十万到几百万条核酸分子同时进行序列测定,从而使得对一个物种的转录组和基因组进行细致全貌的分析成为可能,所以又被称为深度测序(Deep sequencing)。NGS主要的平台有Roche(454 & 454+),Illumina(HiSeq 2000/2500、GA IIx、MiSeq),ABI SOLiD等。 基因:Gene,是遗传的物质基础,是DNA或RNA分子上具有遗传信息的特定核苷酸序列。基因通过复制把遗传信息传递给下一代,使后代出现与亲代相似的性状。 DNA:Deoxyribonucleic acid,脱氧核糖核酸,一个脱氧核苷酸分子由三部分组成:含氮碱基、脱氧核糖、磷酸。脱氧核糖核酸通过3',5'-磷酸二酯键按一定的顺序彼此相连构成长链,即DNA链,DNA链上特定的核苷酸序列包含有生物的遗传信息,是绝大部分生物遗传信息的载体。
DNA甲基化的总结
DNA甲基化是指在DNA甲基转移酶(DNMTs)的催化下,将甲基基团转移到胞嘧啶碱基上的一种修饰方式。它主要发生在富含双核苷酸CpG岛的区域,在人类基因组中有近5万个CpG岛[5]。正常情况下CpG岛是以非甲基化形式(活跃形式)存在的,DNA甲基化可导致基因表达沉默。DNMTs的活性异常与疾病有密切的关系,例如位于染色体上的DNMT3B基因突变可导致ICF综合征。有报道[6]表明,重度女性侵袭性牙周炎的发生与2条X染色体上TMP1基因去甲基化比例增高有关。DNMT基因的过量表达与精神分裂症和情绪障碍等精神疾病的发生也密切相关。风湿性疾病等自身免疫性疾病特别是系统性红斑狼疮(SLE)与DNA甲基化之间关系已经确定[7],在SLE病人的T细胞发现DNMTs活性降低导致的异常低甲基化。启动子区的CpG岛过度甲基化使抑癌基因沉默,基因组总体甲基化水平降低导致一些在正常情况下受到抑制的基因如癌基因被激活[8],都会导致细胞癌变。 甲基化作用是转录水平上表达调控的基本方式之一。由于宿主细胞基因组DNA中不 同位点的甲基化程度存在某种平衡,并形成一定的空间结构特点。一旦转基因的整合破坏了这种平衡及空间特征,破坏后的结构便成为宿主基因组防御系统识别的信号,使新整合的DNA 序列发生不同程度的甲基化,甲基化基因序列则通过抑制甲基化DNA结合蛋白(MeCP2)的结合而抑制转录的顺利进行Ⅲo。在拟南芥中发现了DNA甲基化可以导致基因沉默汹埘]。在基因沉默过程中,外源或内源性信号引起部分DNA序列中CpG的甲基化,甲基化CpG结合域蛋白2(MeCP2)结合到甲基化的胞嘧啶上聚集HDACs使组蛋白去乙酰化,该蛋白与去乙酰化的组蛋白通过聚集更多的DNA 甲基转移酶来加强沉默信号,从而引起基因沉默H?。 ?。DNA甲基化对染色质结构和基因表达的作用很可能是通过一组蛋白介导的,这些蛋白可能含有共同的高度保守的甲基化的CpG结合结构域(MBD)L45 J。DNA甲基化在基因印记、x染色体失活、某些疾病的发生发展中发挥重要作用。其直接作用机制可能是CpG岛甲基化干扰了一些转录因子(transcription factor,TF)与基因调控区的结合,使甲基从DNA分子大沟中突出,从而阻止转录 因子与基因相互作用。间接机制可能是由于甲基化DNA与甲基化DNA结合蛋白结合或DNA甲基化改变染色质结构,这2种情况都间接阻碍TF与DNA结合从而抑制转录m1。DNA甲基化一般是通过转录抑制机制来调节特定基因的,具体的机制可能有:5一MeC伸入DNA双螺旋大沟,影响转录因子的结合;序列特异的甲基化DNA结合蛋白(MDBP一1,MDBP一2)与甲基化的启动子序列特异性结合而抑制转录因子与靶序列的结合;甲基化CpG结合蛋白(MeCPl,MeCP2)与甲基化的二核苷酸CpG结合,发挥类似转录抑制蛋白的作用H“。一般DNA甲基化会通过干扰转录因子与识别位点结合和招募组蛋白乙酰转移酶(histon acefltransfeI"SeS,HATs)、组蛋白去乙酰化酶(histone deacetylases,HDACs)形成辅助阻遏复合体,使基因沉默而抑制其表达,而去甲基化则使沉默的基因重新激活Ⅲ卜 DNA甲基化尤其是基因启动子区CpG岛的高甲基化,会导致基因表达的下降或沉默。甲基化抑制基因的表达目前认为要有两个方面,一方面甲基化引起的基因结构改变可直接阻碍一些转录因子与其结合位的结合;另一方面可能与一些甲基化
DNA甲基化检测技术全攻略
DNA甲基化检测技术全攻略 近年来涌现出不少DNA甲基化的检测技术,少说也有十几种。大致可以分为两类:特异位点的甲基化检测和全基因组的甲基化分析,后者也称为甲基化图谱分析(methylation profiling)。下面大家介绍一些常用的方法。 特异位点的甲基化检测 甲基化特异性PCR(MS-PCR) 这种方法经济实用,无需特殊仪器,因此是目前应用最为广泛的方法。在亚硫酸氢盐处理后,即可开展MS-PCR。在传统的MSP方法中,通常设计两对引物,一对MSP引物扩增经亚硫酸氢盐处理后的DNA模板,而另一对扩增未甲基化片段。若第一对引物能扩增出片段,则说明该检测位点存在甲基化,若第二对引物能扩增出片段,则说明该检测位点不存在甲基化。 这种方法灵敏度高,可用于石蜡包埋样本,且不受内切酶的限制。不过也存在一定的缺陷,你要预先知道待测片段的DNA序列,并设计出好的引物,这至关重要。另外,若存在亚硫酸氢盐处理不完全的情况,那可能导致假阳性。 亚硫酸氢盐处理+测序 这种方法一度被认为是DNA甲基化分析的金标准。它的过程如下:经过亚硫酸氢盐处理后,用PCR扩增目的片段,并对PCR产物进行测序,将序列与未经处理的序列进行比较,判断CpG位点是否发生甲基化。这种方法可靠,且精确度高,能明确目的片段中每一个CpG位点的甲基化状态,但需要大量的克隆测序,过程较为繁琐、昂贵。 联合亚硫酸氢钠的限制性内切酶分析法(COBRA) DNA样本经亚硫酸氢盐处理后,利用PCR扩增。扩增产物纯化后用限制性内切酶(BstUI)消化。若其识别序列中的C发生完全甲基化(5mCG5mCG),则PCR扩增后保留为CGCG,BstU I能够识别并进行切割;若待测序列中,C未发生甲基化,则PCR后转变为TGTG,BstUI识别位点丢失,不能进行切割。这样酶切产物再经电泳分离、探针杂交、扫描定量后即可得出原样本中甲基化的比例。 这种方法相对简单,可快速定量几个已知CpG位点的甲基化,且需要的样本量少。然而,它只能获得特殊酶切位点的甲基化情况,因此检测阴性不能排除样品DNA中存在甲基化的可能。 荧光定量法(Methylight) 此种方法利用TaqMan? 探针和PCR引物来区分甲基化和未甲基化的DNA。首先用亚硫酸氢盐处理DNA片段,并设计一个能与待测位点互补的探针,随后开展实时定量PCR。这种方法最大的优势在于其高通量和高敏感性,且无需在PCR后电泳、杂交等操作,减少了污染和操作误差。 Qiagen就提供了多种预制的MethyLight分析。EpiTect MethyLight PCR Kit包括了两条甲基化敏感的TaqMan探针和2条甲基化不敏感的PCR引物。随着目标序列甲基化状态的不同,只有FAM标记的亚硫酸氢盐转化的甲基化DNA特异的TaqMan探针,或只有VIC
表观遗传学DNA甲基化分析软件
表观遗传学是与遗传学(genetic)相对应的概念。遗传学是指基于基因序列改变所致基因表达水平变化,如基因突变、基因杂合丢失和微卫星不稳定等;而表观遗传学则是指基于非基因序列改变所致基因表达水平变化,如DNA甲基化和染色质构象变化等;表观基因组学(epigenomics)则是在基因组水平上对表观遗传学改变的研究。所谓DNA甲基化是指在DNA 甲基化转移酶的作用下,在基因组CpG二核苷酸的胞嘧啶5'碳位共价键结合一个甲基基团。正常情况下,人类基因组“垃圾”序列的CpG二核苷酸相对稀少,并且总是处于甲基化状态,与之相反,人类基因组中大小为100—1000 bp左右且富含CpG二核苷酸的CpG岛则总是处于未甲基化状态,并且与56%的人类基因组编码基因相关。人类基因组序列草图分析结果表明,人类基因组CpG岛约为28890个,大部分染色体每1 Mb就有5—15个CpG岛,平均值为每Mb含10.5个CpG岛,CpG岛的数目与基因密度有良好的对应关系[9]。由于DNA甲基化与人类发育和肿瘤疾病的密切关系,特别是CpG岛甲基化所致抑癌基因转录失活问题,DNA甲基化已经成为表观遗传学和表观基因组学的重要研究内容。 第一类:基于引物设计功能的软件。此类软件主要是针对重亚硫酸盐序列进行甲基化特异性PCR(methylation-specific PCR, MS-PCR or MSP)和重亚硫酸盐测序(bisulfite sequencing, BS)引物的设计。由于重亚酸盐修饰的特殊性,使常规的分子生物学软件,如Primer Premier、Oligo、DNASis、DNA Max、V ecter NTI Suit等均无法适用于MSP和BS引物的设计。国外的研究者根据MSP和BS的特殊性,分别提供了多种免费或付费的专业软件,其中,某些软件,如MethPrimer已被国内外研究者广泛使用。 软件:MethPrimer 主页:https://www.360docs.net/doc/cb11191339.html,/methprimer 简介:设计甲基化特异性PCR(methylation-specific PCR, MS-PCR or MSP)和重亚硫酸盐测序(bisulfite sequencing)引物的最早和最经典的软件,也是一款免费软件,它广受表观遗传学研究者的喜爱,也是DNA甲基化研究者使用最频繁的一款表观遗传学DNA甲基化研究工具软件。就本人及同事的使用经验而言,该软件所设计的引物扩增效率和特异性均非常好,不过,在实际应用中,如果能配合Primer Premier软件对引物的Tm值进行适当的调整或修改,可进一步提高PCR的扩增效率。此外,此款软件在进行引物设计的过程中,会自动对待分析序列的CpG岛进行分析,它所得到的结果与其它几款CpG岛专业分析软件(见下)一致。当然,如果用户只想对CpG岛的分析和密度进行分析时,可选择其它CpG岛专业分析软件。 文献:Li LC, Dahiya R. MethPrimer: designing primers for methylation PCRs. Bioinformatics. 2002 Nov;18(11):1427-31. 评级:★★★★★ 软件:BiSearch 主页:http://bisearch.enzim.hu/ 简介:与MethPrimer类似的专用于重亚硫酸盐修饰序列相关PCR或测序引物设计的。要使用其功能,需要在其网站注册后使用。由于本人并不是很了解该软件,且并未将其与MethPrimer进行比较过,因此,其功能如何,尚不能断言。 文献:Tusnady GE, Simon I, V aradi A, Aranyi T. BiSearch: primer-design and search tool for PCR on bisulfite-treated genomes. Nucleic Acids Res. 2005 Jan 13;33(1):e9 (PMID: 15653630) 推荐:★★★★★ 软件:BioToolKit300 - Primo MSP 3.4 主页:https://www.360docs.net/doc/cb11191339.html,/ 简介:BioToolKit300是一款基于Java语言的分子生物学软件,支持Windows NT / XP和Mac OSX等操作系统,其试用版仅有60天试用期。该软件是集各种常用分子生物学应用工
中关村华康基因研究院----DNA甲基化测序问题集锦
DNA甲基化(DNA methylation)为DNA化学修饰的一种形式,能在不改变DNA 序列的前提下,改变遗传表观。 DNA甲基化在维持细胞正常功能、传递基因组印记,胚胎发育、肿瘤发生等方面发挥重要作用,目前已经成为表观遗传学和表观基因组学的研究热点。 Roche GS FLX Titanium 、IlluminaSolexa GA IIx和AB SOLID 4均可以进行大规模DNA甲基化测序分析,由于IlluminaSolexa GA IIx测序仪具有数据读取量大、成本低等优势, IlluminaSolexa GA IIx测序仪在DNA甲基化测序方面得到广泛应用。 DNA甲基化测序可在全基因组水平上最大限度的、完整的获取甲基化状态信息和与基因表达调控的多重关系,可高效精确完成全基因组甲基化测序及高分辨DNA 甲基化谱式绘制,并可对发现的靶点区进行甲基化特异性PCR验证。 甲基化测序对样品有什么要求? 答:(1)请提供浓度≥10 ng/μg、总量≥200 ng、OD260/280为1.8~2.2的DNA样品;若单次富集的DNA量不够,建议将2~3次免疫沉淀的DNA合并在一起。(2)样品请置于1.5 ml管中,管上注明样品名称、浓度以及制备时间,管口使用Parafilm封口。在运输前将所有样品管固定于50 ml带盖离心管中,再将50 ml管放在封口袋中。为了防止低浓度样品黏附在离心管壁上,请使用 non-stick tube运输DNA ;建议使用冰袋运输,并且尽量选用较快的邮递方式,以降低运输过程中样品降解的可能性。 MeDIP富集DNA片段的流程? 答:甲基化DNA免疫沉淀法(Methylated DNA immunoprecipitation,MeDIP)是一种高效富集甲基化DNA的方法。主要通过与5mC特异性结合的抗体加入到变性的基因组DNA片段中,从而使甲基化的基因组片段免疫沉淀,形成富集。其流程如下(图1): (1)将基因组DNA超声打断成400-500bp片段; (2)加热变性,使双链DNA片段解链形成单链DNA样品; (3)向变性后的单链DNA样品中加入5’-甲基胞嘧啶抗体; (4)使用亲和层析分离(3)步样品中的甲基化DNA片段的抗体复合物,样品中其余的非甲基化DNA片段被洗脱。纯化得到甲基化DNA片段(MeDNA)。
DNA甲基化详解
提到遗传,我们都已经习惯于这样的概念,即基因组的编码信息存在于ACGT 这四种碱基的排列顺序中。然而,诸如胞嘧啶的甲基化修饰及其分布,组蛋白的乙酰化等,同样影响着表型。这就构成了表观遗传学(epigenetics)的主要研究内容。其实,早在1942年,C.H.Waddinton就提出了表观遗传学的概念,他指出,表观遗传与遗传相对,主要研究基因型和表型的关系。而现在,对于表观遗传学,比较统一的认识是,其研究在没有细胞核DNA序列改变的情况时,基因功能的可逆的可遗传的改变。也就是说,在不改变基因组序列的前提下,通过DNA和组蛋白的修饰等来调控基因表达,其中又以DNA甲基化(DNA methylation)最为常见,成为表观遗传学的重要组成部分。随着人类基因组计划的开展,科学家们开始在基因组水平来研究表观遗传学,逐步形成表观基因组学(epigenomics)。表观基因组学就是要在整个基因组水平来研究表观遗传过程以及与这些过程密切相关的特定基因组区域的识别与鉴定。2000年10月,人类表观基因组协会(Human Epigenome Consortium)由欧盟赞助,启动了旨在于人类6号染色体MHC区域首先做出DNA的甲基化图谱的先导计划(Pilot Project)。该计划顺利完成,引导启动了2003年的人类表观基因组计划(Human Epigenome Project,HEP)。2005年,美国国家卫生院(NIH)下属的国立癌症研究所启动了癌症基因组先导计划。2006年,该所与国立人类基因组研究所一起共同启动癌症基因组计划(Cancer Genome Project)。表观基因组学和DNA甲基化与癌症的研究成为新的热点。本文将简要介绍DNA甲基化与CpG岛,癌症与DNA甲基化,和DNA甲基化的重要检测方法。DNA甲基化与CpG岛:在人类表观遗传学研究中,最常见的就是CpG二核苷酸中胞嘧啶的甲基化修饰。其主要过程是,在CpG甲基化结合蛋白(Methyl-CpG Binding Proteins,MBDs) 和DNA甲基化转移酶(DNA methyltransferases, DNMTs)的作用下,使CpG二核苷酸5’端的胞嘧啶转变成为5’甲基胞嘧啶。在正常人类的DNA中,约有3-6%的胞嘧啶被甲基化。在哺乳动物中,约有50,000,000个CpG二核苷酸,其中70%的被甲基化。而那些可被甲基化的CpG 二核苷酸并非随机的分布于基因组序列中,相反,在基因组的某些区域中,通常是基因的启动子区域,5’端非翻译区和第一个外显子区,CpG 序列密度非常高,超过均值5倍以上,成为鸟嘌呤和胞嘧啶的富集区,称之为CpG岛(CpG Islands, CGIs)。CpG岛的概念最早由Adrian Bird提出,他称之为
全基因组重亚硫酸盐测序和简化代表性重亚硫酸盐测序分析流程
#流程大放送#WGBS和RRBS测序分析流程 介绍 WGBS全称Whole Genome Bisulfite Seuqneicng,即全基因组重亚硫酸盐测序。该方法通过Bisulfite处理,将原基因组中未发生甲基化的C碱基转换成U的同时,保留所有甲基化C 的碱基不发生转变,从而帮助科研人员识别发生甲基化的CpG位点。该种测序技术适用于绘制单碱基分辨率的全基因组DNA甲基化图谱。 RRBS全称Reduced Representation Bisulfite Sequencing,即简化代表性重亚硫酸盐测序。该方法在Bisulfite处理前,使用MspI(该酶的酶切位点为CCGG)酶切对样本进行处理,去除低CG含量DNA片段,从而使用较小的数据量富集到尽可能多的包含CpG位点的DNA片段。 相比于WGBS技术,RRBS是一种准确、高效且经济的DNA甲基化研究方法,通过酶切,并进行Bisulfite测序,该方法在保证DNA甲基化状态检测的高分辨率的同时提升测序数据的高利用率。 该项技术可用于以下研究 1、处于特定时期或特定处理条件下的样本中,研究样本中染色体高精度DNA甲基化模式; 2、比较不同细胞、组织、样本间的高精度DNA甲基化修饰模式的差异; 3、疾病样本中,与疾病发生发展相关的高精度DNA甲基化表观遗传机理研究和相关高精度DNA甲基化位点分子标志的探索性研究。
数据处理和分析流程图 分析结果示例图片展示 示例图1 样本中各区域DNA甲基化水平信息统计和样本间差异DNA甲基化分析结果展示[1] 示例图2 差异DNA甲基化区域内转录因子基序识别[1]
示例图3 DNA甲基化水平变化与基因表达水平变化的关联性分析[1] 示例图来源文献 [1]. Ng, C.W., et al., Extensive changes in DNA methylation are associated with expression of mutant huntingtin. Proc Natl Acad Sci U S A, 2013. 110(6): p. 2354-9.
人全基因组甲基化测序项目结题报告
人全基因组甲基化测序项目 结题报告 成都生命基线科技有限公司
目录 一、分析方法 (3) 1.1 全基因组甲基化测序 (3) 1.2 生物信息分析概述 (3) 1.3 数据过滤 (3) 1.4 序列比对 (3) 1.5 甲基化水平 (3) 1.6 DMR检测 (4) 1.7 甲基化水平程度差异 (4) 1.8 GO注释 (4) 1.9 KEGG通路富集 (4) 二、项目流程 (5) 2.1 实验流程 (5) 2.2 信息分析流程 (5) 三、项目结果报告 (6) 3.1 数据基本处理与质控 (6) 3.2 全基因组甲基化水平分析 (8) 3.3 甲基化C碱基中CG, CHG 与CHH的分布比例 (9) 3.4 甲基化CG、CHG和CHH的甲基化水平分布 (10) 3.5 甲基化的CG,CHG,CHH附近碱基的序列特征分析 (10) 3.6 染色体水平的甲基化C碱基密度分布 (11) 3.7 基因组的不同区域的甲基化分布特征 (11) 3.8 基因组不同转录元件中的DNA平均甲基化水平 (12) 3.9 DMR的检测 (12) 3.10 DMR相关基因的GO和Pathway分析 (14) 四、参考文献 (15)
一、分析方法 1.1 全基因组甲基化测序 首先采用Covaris聚焦超声仪对合格的DNA样品进行打断。加入End Repair Mix置于20℃ 30分钟进行末端修复后,用QIA quick PCR Purification Kit(Qiagen)纯化DNA片段。使用A-Tailing Mix置于37℃ 30分钟在3’末端加A碱基,然后在DNA片段两端连接上测序接头。采用EZ DNA Methylation-Gold kit(ZYMO)进行Bisulfite处理,使用2%琼脂糖凝胶进行片段选择,并使用QIA quick Gel Extraction kit (QIAGEN)回收目标片段。最后使用Agilent 2100 Bioanaylzer和ABI StepOnePlus Real-Time PCR System对样品文库进行质控与定量。合格文库采用Illumina平台进行测序。 1.2 生物信息分析概述 得到下机数据后,首先进行数据过滤,去掉低质量数据,得到可用数据。完成数据过滤后,需检测可用数据量是否符合合同要求。检测合格后,将可用数据与参考基因组进行比对,得到比对结果。在确认比对质量合格后,使用唯一比对数据计算得到全基因组C碱基甲基化信息,进行信息分析处理,得到标准信息分析结果和个性化分析结果。 1.3 数据过滤 数据过滤包括去、污染以及低质量序列。数据过滤分析使用华自主的分析软件,低质量的reads包括以下两类,符合任意一条的都会被剔除: 1) N > 10%; 2) 质量值小于20的碱基>10%。 完成过滤后的reads称为clean reads,这些数据存储为FASTQ格式(参见帮助页中的FASTQ格式)。 1.4 序列比对 过滤完成后,clean data与参考基因组进行比对(BSMAP),并计算每个样品的比对率和bisulfite转化率等统计信息。 1.5 甲基化水平 甲基化水平是支持甲基化的reads数占所有覆盖该位点的reads数的比例[3]。计算公式如下: Nm为改为点是甲基化C的reads数,Nnm为该位点是非甲基化C的reads数。
m6A 甲基化研究方法
RNA甲基化修饰(m6A)研究思路及方案设计 RNA甲基化修饰约占所有RNA修饰的60%以上,而N6-甲基腺嘌呤(N6-methyladenosine, m6A)是高等生物mRNA和lncRNAs上最为普遍的修饰。目前发现microRNA,circRNA, rRNA, tRNA 和snoRNA上都有发生m6A修饰。m6A修饰主要发生在RRACH序列中的腺嘌呤上,其功能由“编码器(Writer)”、“消码器(Eraser)”和“读码器(Reader)”决定[1]。“编码器(Writer)”即甲基转移酶,目前已知这个复合物的成分有METTL3,METTL14, WTAP和KIAA1429;而ALKBH5和FTO作为去甲基酶(消码器)可逆转甲基化;m6A由m6A结合蛋白识别,目前发现m6A结合蛋白(读码器)有YTH结构域蛋白(包括YTHDF1, YTHDF2, YTHDF3,YTHDC1和YTHDC2)和核不均一蛋白HNRNP家族(HNRNPA2B1和HNRNPC)。 m6A酶系统 METTL3是早先被鉴定为结合SAM的组件,其缺失引起小鼠胚胎干细胞、Hela细胞和HepG2 细胞中m6A peaks的减少。METTL3及其同源蛋白METTL14定位在富含剪切因子的细胞核内亚细胞器-核小斑(Nuclear speckle)上,显示m6A修饰可能和RNA的剪切加工相关。WTAP与METTL3–METTL14 二聚体相互作用,并共定位于核小斑,影响甲基化效率,参与mRNA剪。而KIAA1429作为候选的甲基转移酶复合体的新亚基,是整体甲基化进程所必须的[2]。FTO是ALKB家族的成员,作为第一个被发现的去甲基酶,可影响剪切因子SRSF2的RNA结合能力,进而调控pre-mRNA的剪切加工过程[3]。目前已发现FTO调节异常与肥胖、大脑畸形和生长迟缓相关,揭示m6A可能对这些疾病具有重要的调节功能[4-6]。ALKBH5是ALKB家族中被发现具有去甲基作用的另一个成员,以RNase A敏感的方式与核小斑共定位,它可直接催化m6A-甲基化腺苷去除甲基而不同于
甲基化测序word版
DNA甲基化检测实验 一、重亚硫酸盐的测序法实验流程(BSP) (Bisulfite Genomic Sequence) 原理:结合重亚硫酸盐的测序法是一种灵敏的能直接检测分析基因组DNA甲基化模式的方法。重亚硫酸盐处理后,用针对改变后的DNA序列设计特异性引物并进行聚合酶链式反应(PCR)。PCR产物中原先非甲基化的胞嘧啶位点被胸腺嘧啶所替代,而甲基化的胞嘧啶位点保持不变。PCR产物克隆后进行测序。通过这个方法能得到特定位点在各个基因组DNA分子中的甲基化状态。该方法特点是: ?特异性高,它能够提供特异性很高的分析结果,这是所有其他研究甲基化的分析方法所不能比拟的; ?灵敏度高,可以用于分析少于100个细胞的检测样品。用微量的基因组DNA进行分析就能得到各个DNA分子精确的甲基化位点分布图。 重亚硫酸盐测序法技术实验流程 A. DNA制备 用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。 B.重亚硫酸盐处理 C. DNA纯化 用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。 D. PCR扩增 E. PCR产物琼脂糖电泳后回收纯化 F. PCR产物连接到pMD19-T (Takara) 载体中克隆及测序。 G.用分析软件对各样本测序结果进行甲基化程度分析 二、甲基化特异性的PCR实验流程 (methylation-specific PCR, MSP) 原理:甲基化特异性的PCR是一种灵敏度高且操作相对简单的甲基化研究方法。重亚硫酸盐
DNA 甲基化分析技术
文章编号:1007-4287(2005)02-0304-03 DNA 甲基化分析技术 黄 庆,府伟灵3 (第三军医大学西南医院检验科,四川重庆400038) 基金项目:国家自然科学基金资助项目(编号:30370398);重庆市国家自然科学基金资助项目(编号:2004227222);第三军医大学科研基金资助项目(编号:XG 200326)3通讯作者 甲基化胞嘧啶(5’2methylated cytosine ,5mC )被称之为人类DNA 的第5种碱基。肿瘤表观基因组 (cancer epigenomics )概念的提出和人类表观基因组计划(Human E pigenetic Project ,HEP )的实施将表观遗传学研究又推向了一个新的高度[1]。了解DNA 甲基化分析技术的原理、适应范围和优缺点有利于研究者根据自身不同需求和设备条件来选择有效方法达到最终目的[2,3]。1 DNA 甲基化非特异性分析 属于早期的DNA 甲基化分析技术,其主要特征是只能分析基因组水平的5mC 比例,主要包括高效液相色谱、高效毛细管电泳、薄层层析、M.Sss I 甲基转移酶分析、去水乙缩氯醛反应、5mC 免疫学抗体技术等[2,3]。随着人类基因组计划的完成及HEP 的实施,单纯地判断基因组5mC 水平已经不能满足现代表观遗传学研究的需求。2 DNA 甲基化特异性分析是一类可分析特定基因或序列位点甲基化状态的分析技术,也是近年来发展比较迅速的技术之一。根据其作用原理,可将其分为依赖和不依赖于重亚硫酸盐修饰的检测技术。211 不依赖于重亚硫酸盐修饰的检测技术 主要包括肼反应、高锰酸反应和限制性内切酶技术[2,3] 。其中,前两种技术因此操作费时和步骤复杂等原因已很少被研究者所采用。限制性内切酶技术是碱基未经任何修饰的情况下对基因组或已知基因进行甲基化分析的最常用方法之一,其原理是基于甲基化敏感限制性核酸内切酶(methylation 2sensitive restric 2tion endonucleases ,MS 2RE )和甲基化不敏感限制性核酸内切酶(methylation 2sensitive restriction endonucleas 2es ,MIS 2RE )的单酶切/组合酶切和随后的S outhern 杂交或PCR 检测酶切产物属性而确定DNA 甲基化 状态和模式。将S outhern 杂交和PCR 技术引入酶切产物的后续分析大大提高了检测灵敏度,特别是当DNA 数量有限时若采用PCR 技术可大大节约DNA 的用量,此外,PCR 技术的引入还可对甲基化DNA 进行定量分析,如固相定量引物延伸方法(s olid 2phase quantitative primer extension method )、竞争性引 物结合位点(com petitive primer binding sites )技术、连接物引导PCR (ligation 2mediated PCR ,LM 2PCR )和单核苷引物延伸反应等[2~4]。不过,由于限制酶自身的特性使该技术只能判断酶切位点所在序列的甲基化状态,因此,如果使用了不同的酶切体系可导致不同研究者之间的结果存在差异而缺乏比较性。212 依赖于重亚硫酸盐修饰的检测技术 是指利 用重亚硫酸盐对胞嘧啶和5mC 修饰的差异而达到鉴别二者的一类技术,重亚硫酸钠修饰后胞嘧啶被转变成尿嘧啶,而5mC 则在修饰前后保持不变。因此,利用DNA 测序技术或PCR 技术等可达到对5mC 的特异性分析,主要包括重亚硫酸测序、甲基化特异性PCR (methylation 2specific PCR ,MS 2PCR )、甲基化特异性单核苷引物延伸(methylation 2sensitive single nu 2cleotide primer extension ,Ms 2S NuPE )、重亚硫酸盐限制酶组合分析(combined bisulphate restriction analysis ,C OBRA )、甲基化敏感差异展示分析(methylation 2sen 2sitive 2representation difference analysis ,MS 2RDA )和甲 基化敏感单链构象分析(methylation 2sensitive single 2strand con formation analysis ,MS 2SCC A )等[5~11]。 MS 2PCR 是目前研究DNA 甲基化最为常用的方 法之一,其中,引物设计是其成败的关键[2,6]。除标准的MS 2PCR 之外,荧光MS 2PCR 和定量MS 2PCR 等新方法将标准MS 2PCR 的检测灵敏度和适用性又推向了一个新的高度[7]。尽管如此,由于MS 2PCR 引物设计的特殊性使其仅能判断引物序列所在C p G 位点的甲基化状态,因此,不同的MS 2PCR 引物可能会产生不同的分析结果。Ms 2S NuPE 将基因突变检测和等位基因特异性表达分析中所应用的单核苷引物延伸技术引用入DNA 甲基化分析,其特点是可同 — 403—Chin J Lab Diagn ,April ,2005,V ol 9,N o.2
甲基化位点
[试验相关] 表观遗传学(Epigenetics)之DNA甲基化 DNA甲基化是表观遗传学(Epigenetics)的重要组成部分,在维持正常细胞功能、遗传印记、胚胎发育以及人类肿瘤发生中起着重要作用,是目前新的研究热点之一。随着对甲基化研究的不断深入,各种各样甲基化检测方法被开发出来以满足不同类型研究的要求。这些方法概括起来可分为三类:基因组整体水平的甲基化检测、基因特异位点甲基化的检测和新甲基化位点的寻找。 近15年来,人们越来越认识到DNA甲基化研究的重要性,开发出一系列检测DNA的方法。根据研究目的这些方法分为:基因组整体水平的甲基化检测,特异位点甲基化的检测和新甲基化位点的寻找。根据研究所用处理方法不同可以分为:基于PCR的甲基化分析方法;基于限制性内切酶的甲基化分析方法;基于重亚硫酸盐的甲基化分析方法和柱层法等。DNA甲基化的分析方法很多,可分为总基因组甲基化的检测和单基因序列特异性甲基化分析的研究。总基因组甲基化的检测又分为全基因组序列特异性甲基化分析和基因组非特异性甲基化水平的研究。前者包括甲基化差异性杂交显示(differential methylation hybridization,DMH)、寡核苷酸微阵列法和基因组限制性酶切扫描法(restriction landmarkgenomescanning,RLGS);后者包括3H—SAM掺人后液闪检测法和高压液相色谱法。 对单基因序列特异性甲基化分析包括传统的甲基化敏感的限制性内切酶(methylation sensitive restriction endonucleases,MSREs)分析、比较简洁的甲基化特异性 PCR(methylation specific PCR,MSP)、全面反映甲基化情况的亚硫酸氢钠变性后测序(bisulfitegenomic sequencing)、甲基化敏感性单核苷酸引物扩增(methylation sensitive single nucleotide primer extension,Ms—SnuPE)、较新颖的甲基化荧光检测(methylight)、结合亚硫酸氢钠变性的限制性酶分析(combined bisulfite restrictionan alysis,COBRA)、酶的区域性甲基化特异性分析(enzymatic regional methylation assay,ERMA)和变性高压液相色谱法(denaturing high performance liquid chromatography,DHPLC)。 甲基化敏感的单核苷酸的扩增(Ms—SnuPE) Ms—SnuPE即甲基化特异的单核苷酸扩增,它能对不同甲基化特异位点进行快速定量,是一种快速估计特异性CpG位点甲基化不同情况的定量方法。 先用重亚硫酸盐处理基因组DNA,未甲基化的胞嘧啶全部转化为尿嘧啶,而甲基化的胞嘧啶不变。进行PCR扩增,然后取等量扩增产物置于2管中,分别作为Ms—SnuPE单核苷酸引物延伸的模板。设计用于Ms—SnuPE延伸的引物的3’端紧邻待测碱基。同时于2个反应体系中加入等量的Taq酶、引物、同位素标记的dCTP或dTTP。这样,如果待测位点被甲基化,则同位素标记的dCTP会在反应延伸时连于引物末端;若是未被甲基化,则标记的dTTP参与反应。末端延伸产物经电泳分离和放射活性测定后可得出C/T值,即为甲基化与非甲基
